原创内容,欢迎转载,转载请注明出处
主笔:于浩
注意事项
- 蛋白的结构信息预测的方法有很多种,这里每种分析只提供一种方法。我们在实际分析的时候可以选用一种方法,通常这也是很多文章选用的方法。如果预测的结果非常重要,也可以通过不同的数据库来分析,最终整合结果使用。当然,最重要的还是科学问题和生物学意义本身,所以只要能够实现预期目标即可。
- 很多网站可以提供单个蛋白的分析,也可以提供批量分析,但是批量分析的结果并不友好,如果我们希望得到自己想要的结果通常需要自己重新对结果进行分析。比如说用excel配合excel的一些脚本来实现,任何一门语言都可以完成这个工作,我们实验室大部分是通过python脚本来整理的。
- 深度学习已经被用于了蛋白分析,比如说下面介绍的DeepTMHMM,随着AI的持续发展相信以后会有越来越多的基于深度学习的预测工具的出现。
- 如果出现了网站链接失效的话可以去百度上寻找其他的工具或者最新网址。
一、蛋白跨膜区预测
利用TMHMM - 2.0在线预测跨膜区
如果只有几个蛋白,直接用在线工具进行分析就行,目前使用最多的还是TMHMM2.0,所以我们也选用这个就行(关键是科学问题,不是技术)。
1、利用TMHMM2.0分析
首先要打开TMHMM-2.0在线预测网站,在文本框中输入蛋白的序列,输入完成后,其他参数不需要修改,点击“Submit”,开始进行分析。速度很快。
2、结果展示
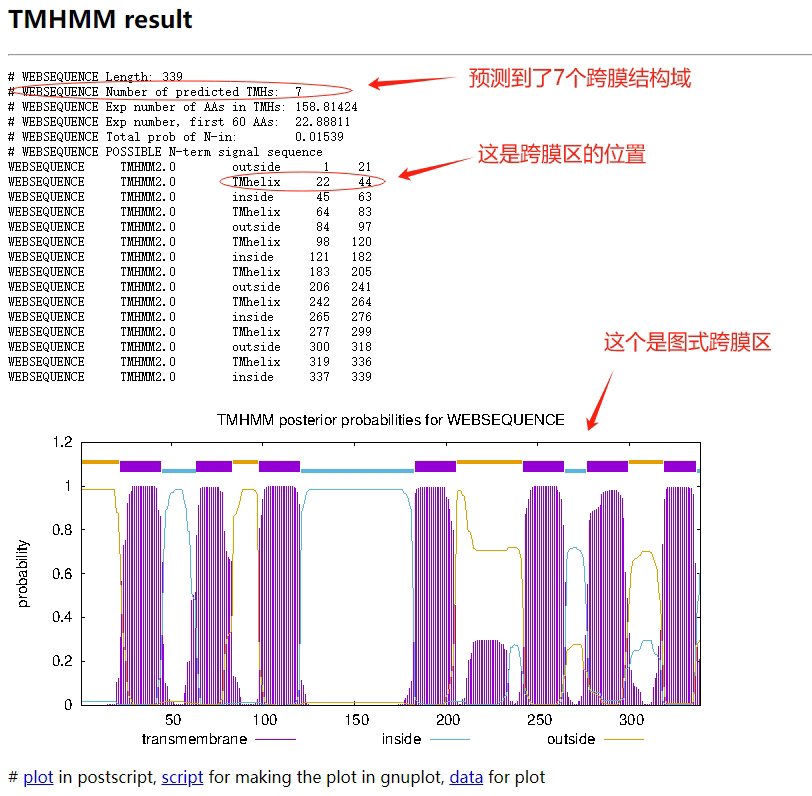
网页生成的结果如下,查看是否有跨膜区就看“Number of predicted TMHs“的数量就行,如果是0就代表了没有跨膜区。
我们分析的这个蛋白预测出来有7个跨膜区,通下面的图像上面来看还有一个区域也有可能是跨膜区,但是根据算法最终并未被预测为跨膜区。
利用TMHMM-2.0在本地进行蛋白跨膜区预测
如果要批量预测蛋白的跨膜结构,那么就要使用本地的TMHMM工具进行了。
1、安装TMHMM
到TMHMM网站的主页,也就是上面的那个地址,在Download下载Linux版本的软件,需要输入邮箱。
# 解压缩
tar -zxvf tmhmm-2.0c.Linux.tar.gz
修改bin文件中的tmhmm和tmhmmformat.pl文件
将第一行的 “#!/usr/local/bin/perl” 和 “#!/usr/local/bin/perl -w”
修改为:
“#!/usr/bin/perl”
将bin文件添加到环境变量中,就可以使用tmhmm了。
2、利用tmhmm预测蛋白的跨膜区
把蛋白序列保存到 ”proteome.fa“ 文件中。
# 结果没有图片,把结果都保存在一个文本文档中,便于后面统一提取信息。
tmhmm -noplot proteome.fa > tmhmm.txt
结果都保存在tmhmm.txt文件中,可以写脚本提取其中的有效信息。
3、批量运行每个蛋白都有单独的结果(有图片)
tmhmm proteome.fa
DeepTMHMM:基于深度学习的模型进行跨膜区的预测
DeepTMHMM是基于深度学习的蛋白跨膜结构域预测工具,根据官方网址的说法被整合到了pybiolib软件包中。
1、安装DeepTMHMM
pip3 install pybiolib
2、使用DeepTMHMM预测蛋白的跨膜区
把要进行分析的蛋白的序列放到一个文本文档里面,我在这里命名为“input.fa”。
用下面的脚本来运行DeepTMHMM(官方推荐的代码)
biolib run DTU/DeepTMHMM --fasta input.fa
3、结果展示
运行结束后会在文件夹里面生成一个名字叫做 “biolib_results” 的文件夹,里面放着运行的结果。
既可以单个蛋白运行也可以多个蛋白运行,运行的结果都在一个叫做 “deeptmhmm_results.md” 的MarkDown文档里面,小伙伴可以自己去把需要的信息提取出来。单个蛋白运行会生成图片,多个蛋白运行不会(推测要设置其他参数)。
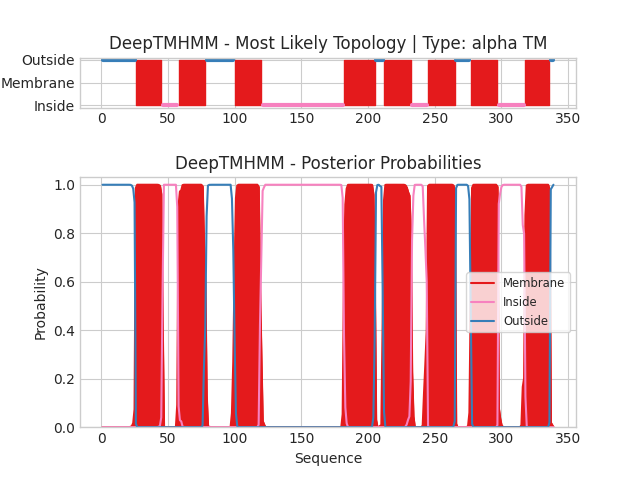
下面是同样的蛋白在DeepTMHMM预测的结果:可以看到结果跟TMHMM 2.0预测的结果不同,DeepTMHMM预测到了8个,上面预测到的那个没有被认为是跨膜区的结构这里被预测为了跨膜区。所以说TMHMM和DeepTMHMM还是有差异的。
究竟选择哪个工具自己根据实际需求选择。(我建议选择TMHMM 2.0就行)
用于分析的蛋白质的序列:A0A067NGT8
MDTDPVADEINCGSGGGADTLFGLRVASIFIILVGSTAGALFPVLARRSSWLHVPKSMFDFAKYFGSGVIIATAFIHLLDPALEALGSECLAPGWSEYPYALALCLLSIFMIFIVELVAFRWGTAKLAKLGLRHDPHGHNVGSHVSHGPEGELINEQEKTKADVESQSSGNPMYTESAMAQIIGVAILEFGVVLHSVLIGMTLAVDEDFKVLLVVLVFHQTFEGLGVGSRLAFLQLSPTYNWVPLFGAILYGLTTPIGIAAGLGIRTTYNPGSTTASIVSGVMDALSAGILIYTGLVELLAHDFLFNKEMMNASNAKLAYALGSMLLGCGLMALLGKWA
二、蛋白信号肽预测
利用SignalP - 5.0在线预测跨膜区
官方网站
https://services.healthtech.dtu.dk/service.php?SignalP-5.0
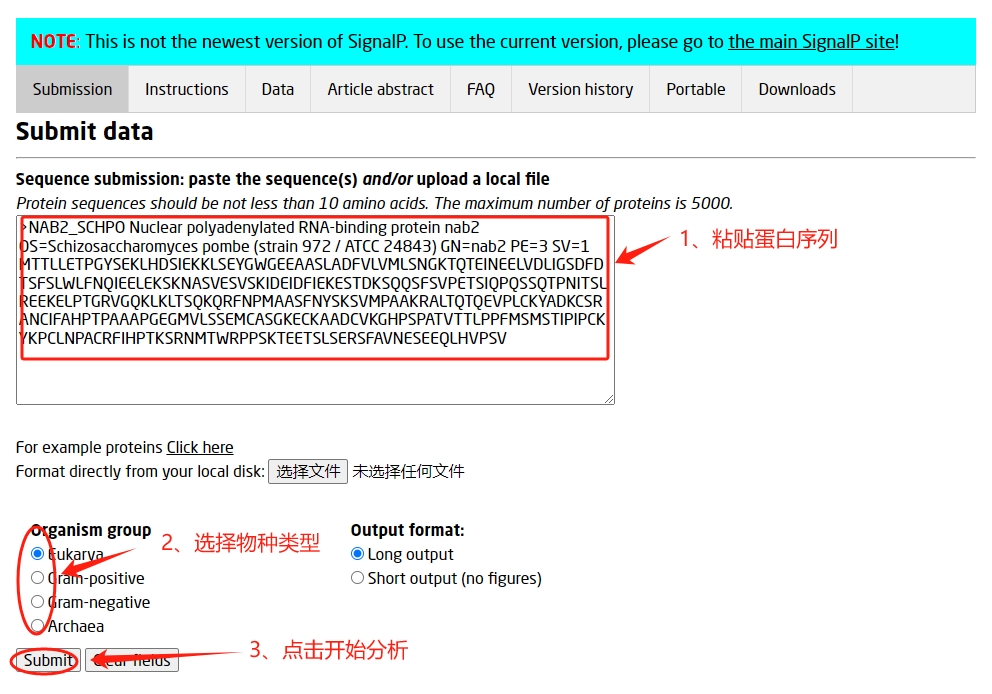
1、利用SignalP - 5.0分析
进入网页地址,将蛋白序列粘贴到文本框中,选择物种分类(默认为真核生物),点击”Submit“开始分析。
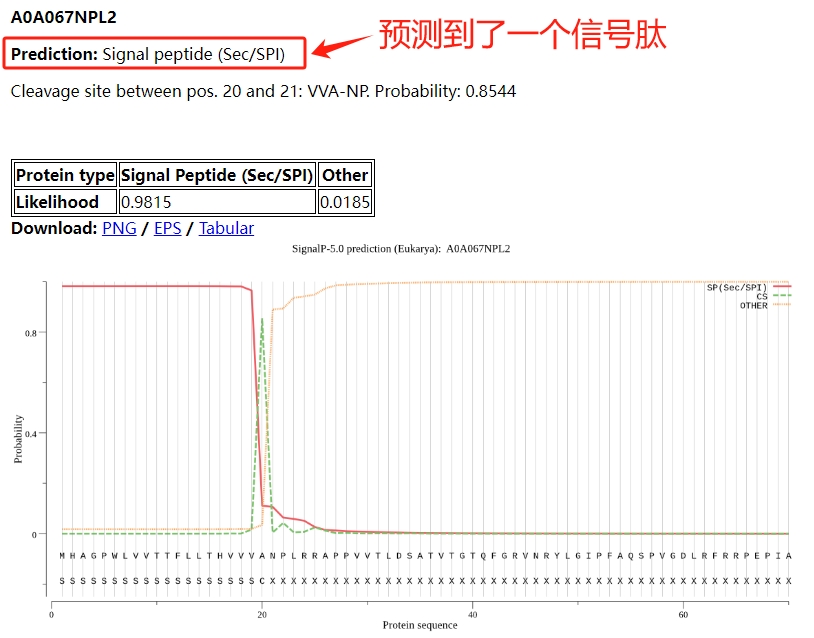
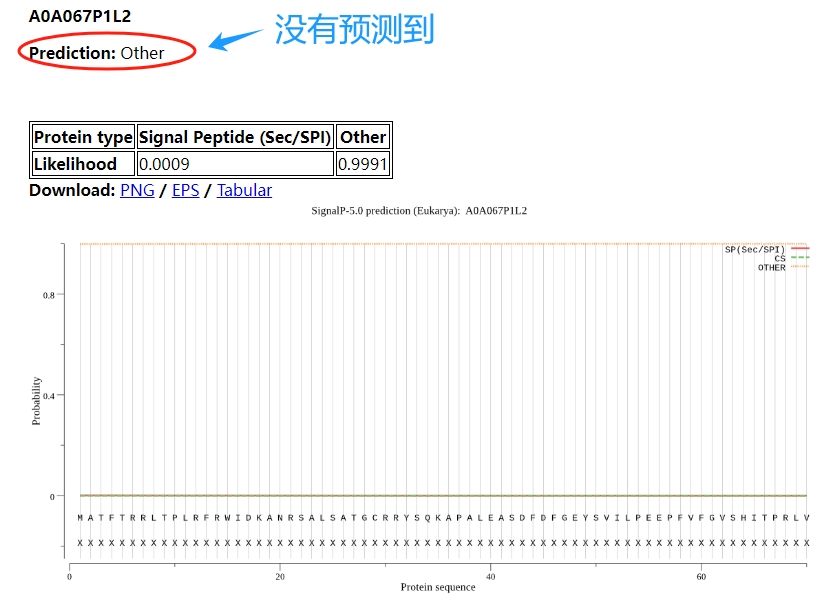
2、结果展示
结果如下,如果预测到了会显示出有信号肽。没有则显示“Other”。
利用服务器本地预测跨膜区
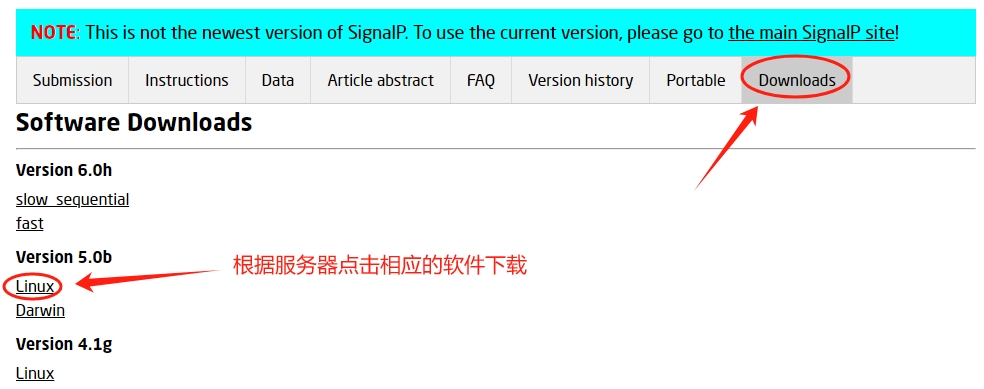
1、安装本地的SignalP
在官网的“Download”菜单中选择想要下载的本地压缩文件,下载后解压后添加环境变量就可以使用。
2、利用本地SignalP来预测信号肽
把蛋白序列保存到 ”proteome.fa“ 文件中。
# 如果只要一个表格样式的结果,就用下面的代码运行
signalp -fasta proteome.fa
# 如果想要生成图片,生成细节,则需要加上long这个参数。
signalp -fasta proteome.fa -format long
蛋白二级结构预测
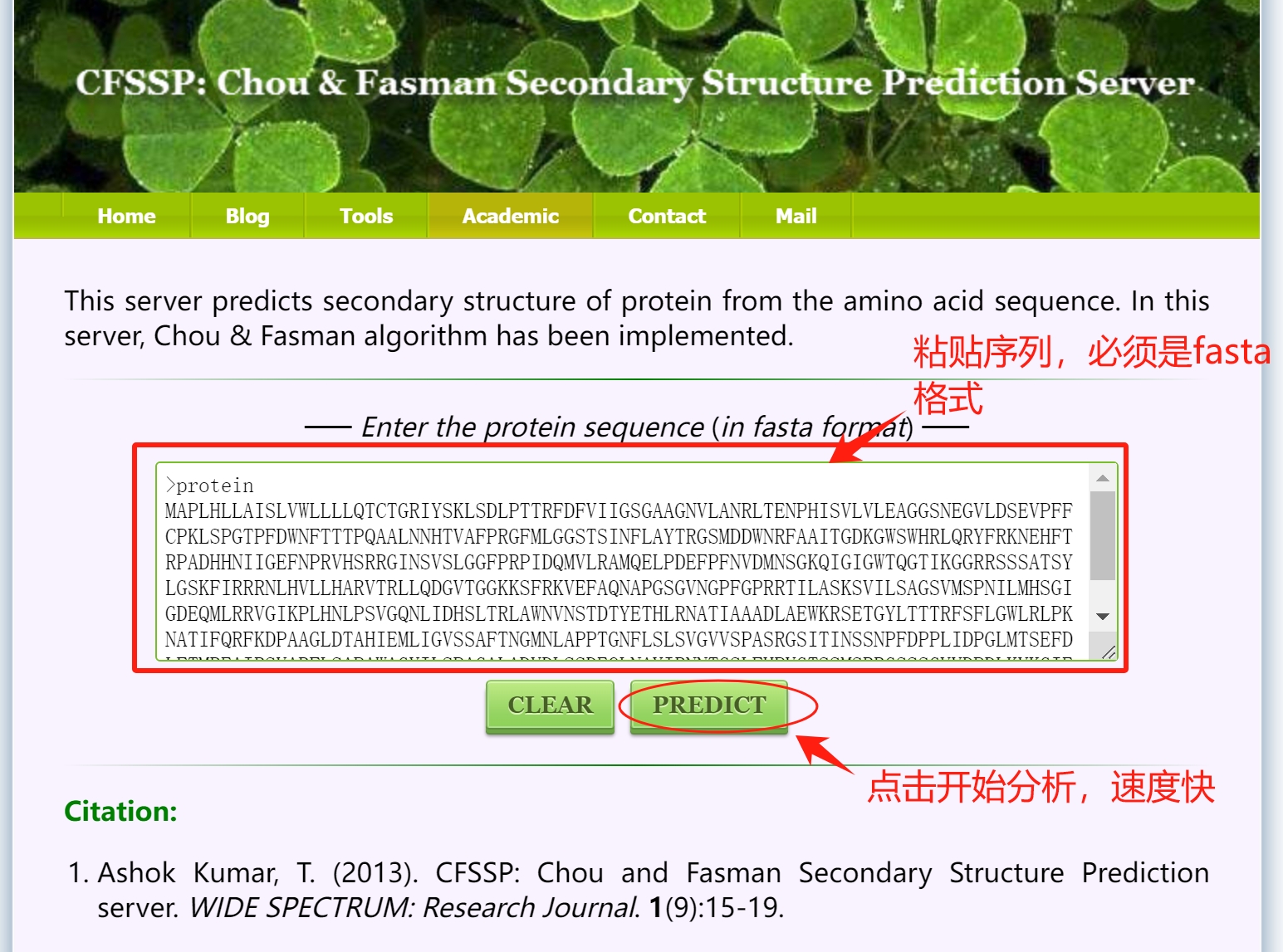
CFSSP
1、二级结构预测
点击进入网站:https://www.biogem.org/tool/chou-fasman/
在文本框中输入蛋白序列,必须是fasta格式,点击“PRIDICT”开始分析,结果秒出。
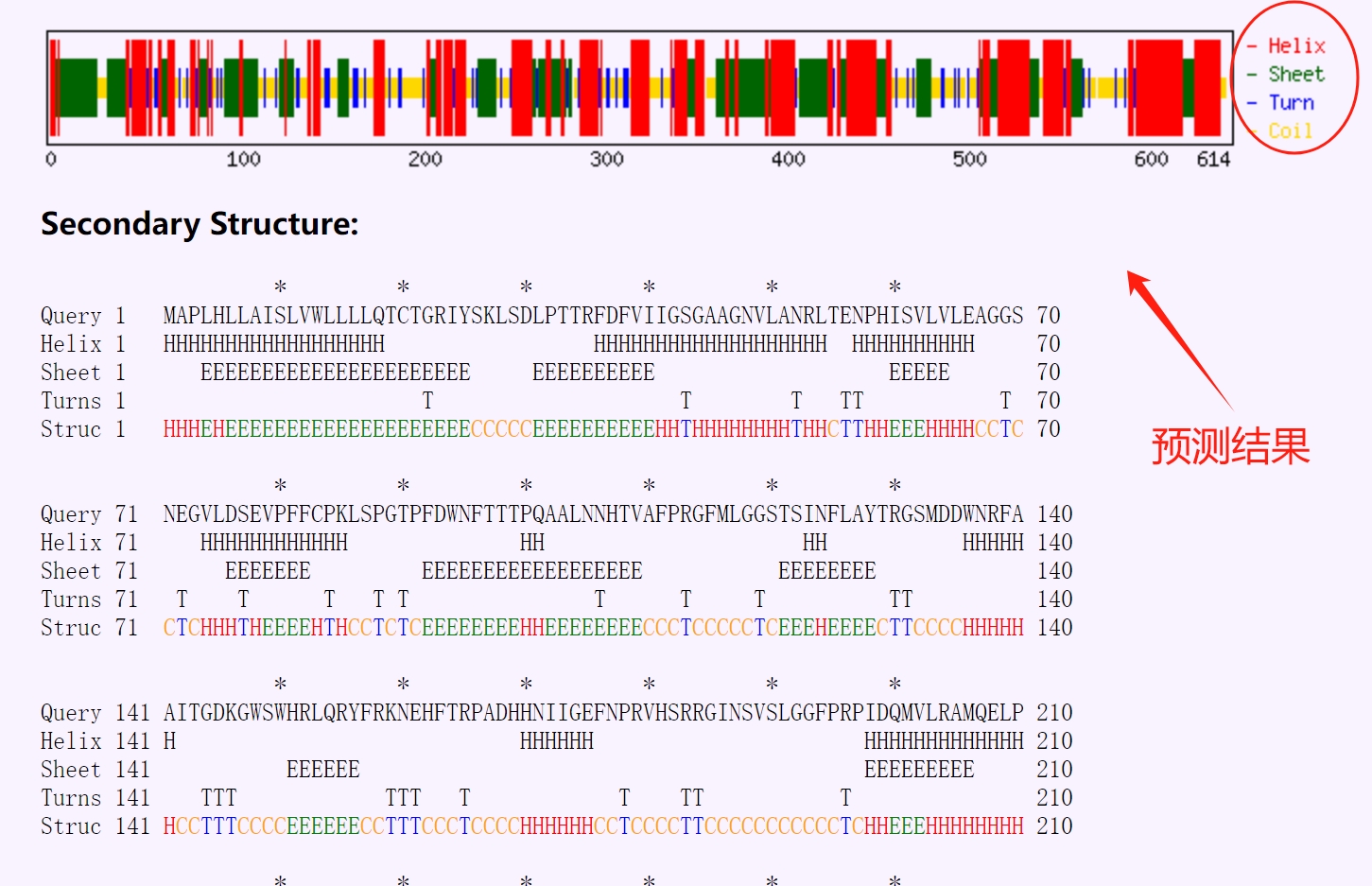
2、结果展示
如下图
PSIPRED
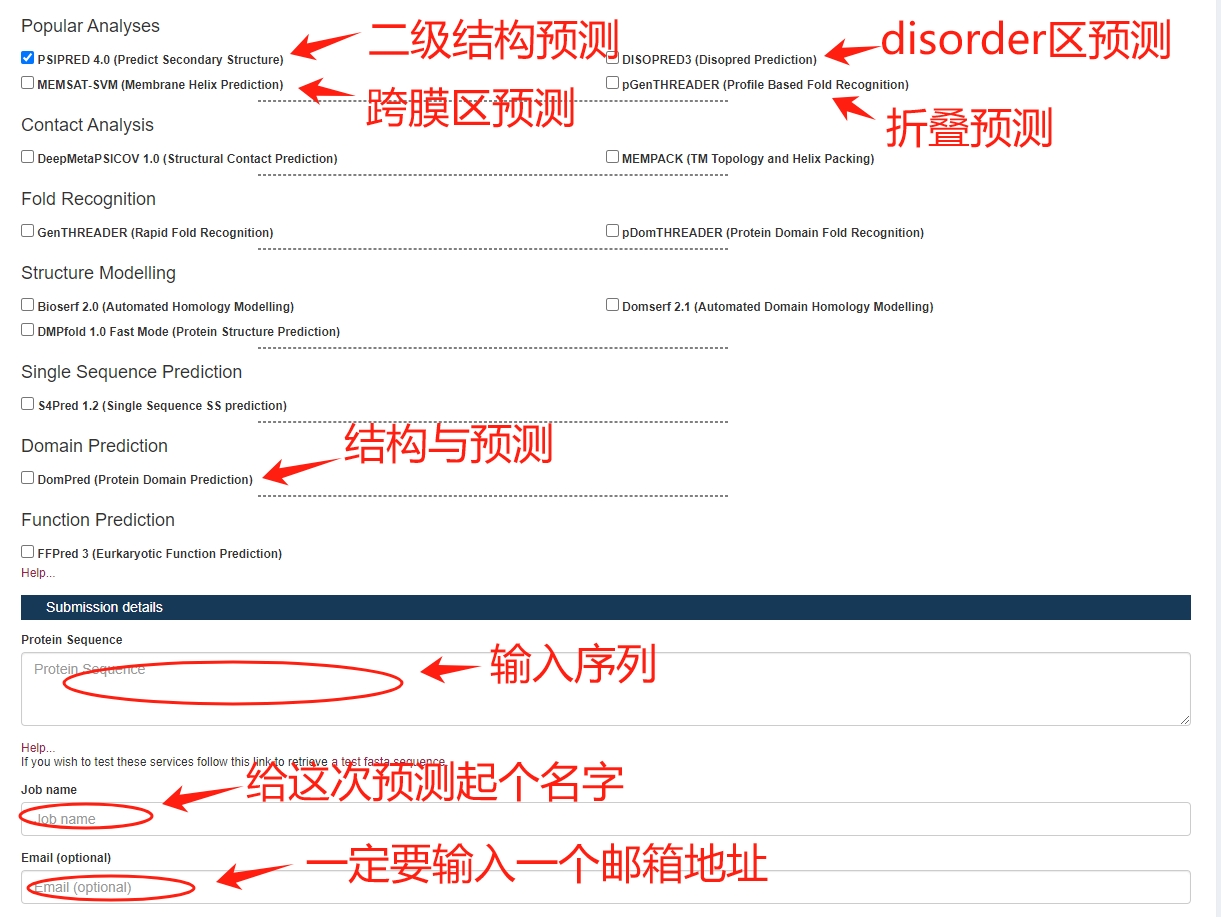
1、序列比对
打开网站:http://bioinf.cs.ucl.ac.uk/psipred/
这个工具预测的慢,所以我们就使用默认参数“预测二级结构”就行,其他不需要勾选。
把蛋白序列粘贴到文本框里面,随便起一个高大上的名字,留下自己的邮箱。(邮箱不是必选项,但是因为这个服务器工作特别慢,2个小时能出结果,所以留邮箱的好处是比对完成会把结果链接网址发到邮箱里面)数据保留一个月。
最后点击“Submit”开始分析。
2、结果展示
也可以记住结果运行中的这个地址,也能够查看结果文件。
结果自己查看就行,这里不展示了。
蛋白亚细胞定位预测
分析工具介绍
跟前面的预测过程一样,很多工具可以进行亚细胞定位的预测,我们可以只选用其中一个的结果,也可以选用多个结果。
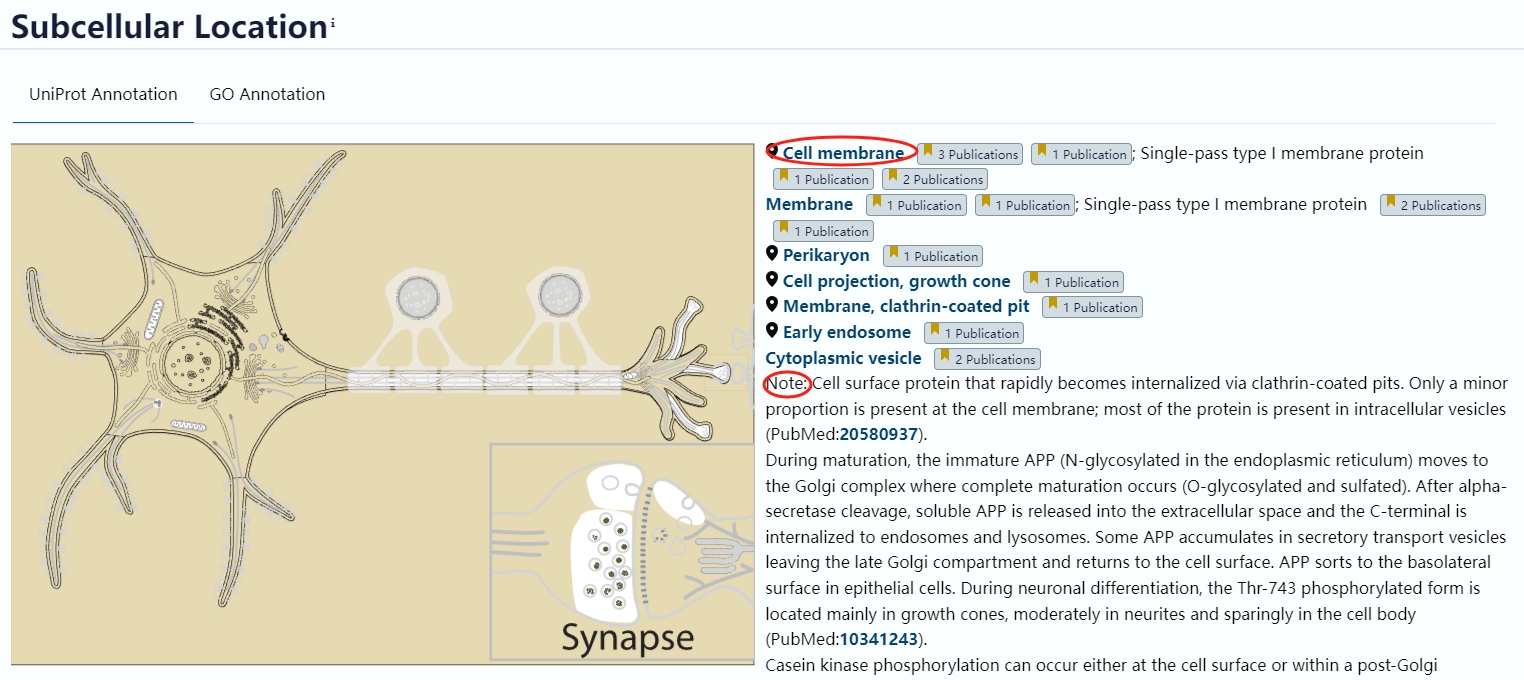
如果这个蛋白可以从UniProt上面找到,那么UniProt上面也同时会给出亚细胞定位,这个时候可以采用UniProt上面的结果,具体结果如下:
常用的亚细胞定位注释分析的网站如下:
WoLF PSORT:https://www.genscript.com/wolf-psort.html
Ilco:http://www.jci-bioinfo.cn
Cell-PLoc: http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc/
CELLO :http://cello.life.nctu.edu.tw/
SherLoc2:http://abi.inf.uni-tuebingen.de/Services/SherLoc2
Predotar:http://urgi.versailles.inra.fr/predotar/predotar.html
uniport :https://www.uniprot.org/
YLoc:https://abi-services.cs.uni-tuebingen.de/yloc/webloc.cgi (测试的时候地址不可用)
DeelLoc 2.0
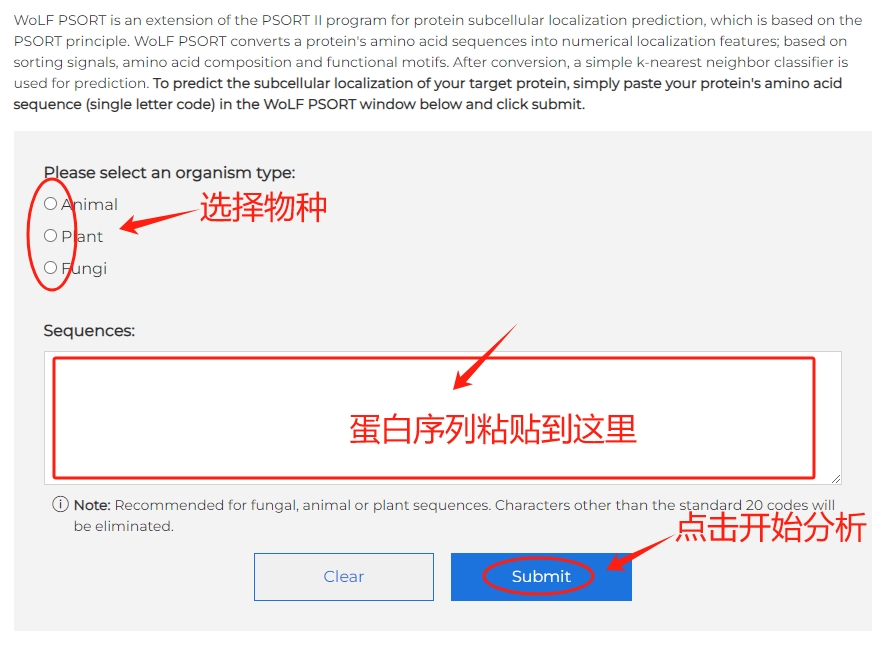
利用WoLF PSORT进行亚细胞定位分析
1、分析流程
WoLF PSORT有两个网址可以进入,分析结果基本一致
第1个网址:https://www.genscript.com/wolf-psort.html
第2个网址:https://wolfpsort.hgc.jp/
点击进入网站,选择合适的物种,将蛋白粘贴到文本框中,点击“Submit”开始进行分析。
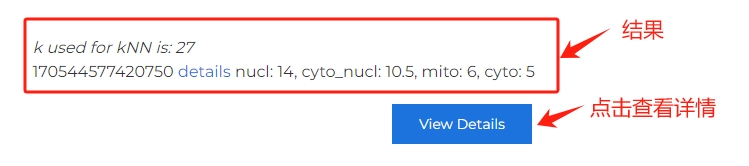
2、预测结果展示
预测的结果如下:
第1行表示结果是用KNN算法整理的,kNN算法中的k值为27。
第2行表示这个蛋白在各个亚细胞定位的可能性:细胞核得分,14,细胞质-细胞核得分10.5,线粒体得分6,细胞质得分5
得分高的可能性高,通常我们选择最高的概率的作为本蛋白的亚细胞定位。
点击“View Details”可以查看一些详细的信息。
下面是细胞位置和缩写的对应表格。
| 描述 | Description | WoLF PSORT site |
|---|---|---|
| 细胞骨架 | cytoskeleton | cysk |
| 细胞质 | cytosol | cyto |
| 内质网 | Endoplasmic reticulum | E.R. |
| 细胞外 | extracelllular | extr |
| 细胞壁 | Cell wall | |
| 高尔基体 | Golgi apparatus | Golg |
| 线粒体 | mitochondrion | mito |
| 细胞核 | nucleus | nucl |
| 细胞质-细胞核 | cytosol nucleus | cyto-nucl |
| 原生质膜,细胞膜 | plasma membrane | plas |
| 过氧化物酶体 | peroxisome | pero |
| 血管膜 | vascular membrane | vacu |
| 叶绿体 | chloroplast | chlo |
| thylakoid lumem |




评论区