原创内容,欢迎转载,转载请注明出处
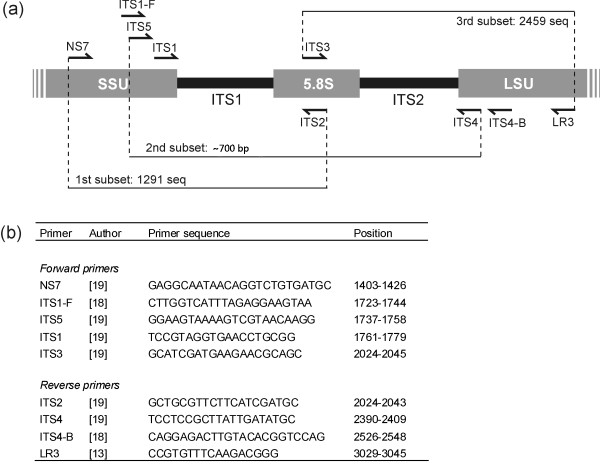
ITS序列是内源转录间隔区(Internally Transcribed Spacer),位于真菌18S、5.8S和28S rRNA基因之间,分别为ITS 1和ITS2。
在真菌中,5.8S、18S和28S rRNA基因具有较高的保守性,而ITS由于承受较小的自然选择压力,在进化过程中能够容忍更多的变异,在绝大多数真核生物中表现出极为广泛的序列多态性。同时,ITS的保守型表现为种内相对一致,种间差异较明显,能够反映出种属间,甚至菌株间的差异。并且ITS序列片段较小(ITS 1和ITS 2长度分别为350 bp和400 bp),易于分析,目前已被广泛用于真菌不同种属的系统发育分析。
其他用于分类的分子标记
translation elongation factor 1-α(TEF1)也是一个常用的用于分类的基因,从文献中查到的引物如下:
| 引物F | 引物F序列 | 引物R | 引物R序列 |
|---|---|---|---|
| TEF1-EF1 | ATGGGTAAGGARGACAAGAC | TEF1-EF2 | GGARGTACCAGTSATCATGTT |
| TEF1-Fa | TCGTCATCGGCCACGTCGACTCT | TEF1-Ra | CAATGACGGTGACATAGTAGCG |
| TEF1-Fa+7 | AACGTCGTCGTCATCGGCCACGTCGACTCT | TEF1-Ra+6 | ACATACCAATGACGGTGACATAGTAGCG |
| TEF1-DermF | CACATTAACTTGGTCGTTATCG | TEF1-DermR | CATCCTTGGAGATACCAGC |
rpb2 gene也被用于真菌分类当中,从文献中查到的引物如下:
| 引物F | 引物F序列 | 引物R | 引物R序列 |
|---|---|---|---|
| bRPB2-6F | TGGGGYATGGTNTGYCCYGC | bRPB2-7R | GAYTGRTTRTGRTCRGGGAAVGG |
ITS测序分析基本流程
- 利用通用ITS测序引物(ITS1和ITS4)以菌丝作为模板进行菌落PCR扩增,并进行测序。如果能够提取基因组之后再以基因组作为模板进行ITS扩增效果更好。
- 对测序峰图进行分析,并决定是否连接载体进行单克隆测序。【重点!】
- 绝对测序结果进行整理,去除不准确信息。
- 利用软件进行系统进化分析(具体参考“”)
一、ITS序列的PCR扩增
1、在扩增ITS序列的时候建议直接用菌丝进行“食用菌菌丝菌落PCR”,具体方法参考链接内容,使用的引物为ITS1和ITS4,具体的序列可以参考“常用质粒和通用引物列表”,扩增利用Taq酶进行扩增即可,下面简单的列出来了一个PCR扩增方案,具体的方法建议参考其他教程。
| 实验组/μL | 阴性对照/μL | |
|---|---|---|
| 2 x Taq酶 Mix | 12.5 | 12.5 |
| ddH2O | 12 | 12 |
| ITS1引物(10 μM) | 1 | 1 |
| ITS4引物(10 μM) | 1 | 1 |
| Template | 1 | – |
| 总体积 | ~25 | ~25 |
| 步骤 | 温度 | 时间(min) |
|---|---|---|
| 1 | 95℃ | 5 min |
| 2 | 95℃ | 15 s |
| 3 | 58℃ | 20 s |
| 4 to 2(共35个循环) | 72℃ | 40 s |
| 5 | 16℃ | ∞ |
2、ITS扩增的PCR产物进行琼脂糖凝胶电泳。
3、如果电泳条带明亮、单一,跟DNA Marker比较大小在~600 bp,阴性对照无条带(有可能会因为污染等问题造成微弱条带),说明扩增的结果是正确的。
4、将PCR扩增产物送到公司进行测序。
二、ITS测序峰图分析
1、第1种情况:是全部是单峰(两边测不准的区域不算)
这种情况最简单,只要按照下面的数据处理去掉不准确的区域就行了,也是我们最希望看到的情况。
2、第2种情况:存在SNP位点
大部分担子菌食用菌细胞内都存在两个不相容的细胞核,属于异核二聚体,这两个细胞核的ITS序列有可能不同,不同菌株两个核的ITS不同的概率不同。(当然也不能完全排除一个核内多个拷贝的ITS序列不同的情况)这种不同有可能是SNP位点,也有可能是存在InDel位点。如果是单核苷酸的差异,在测序峰图上面就会出现下面的这种情况。
如果整个序列只有一个SNP位点,这种情况相对简单,比如说这里的SNP位点是C/T位点。那么就把ITS由一条变为两条,一条这里是C,记为ITS-a,另一条这里是T,记为ITS-b。
如果出现多个SNP位点,根据单纯的测序肯定测不准(只能猜测其中一条的峰永远高一点,但是这点不能确定),在这种情况下如果要获得准确的两条ITS序列,必须要链接载体进行单克隆的测序(参考步骤五)。
3、第3种情况:出现套峰
如果两个ITS的差异不是由SNP导致的而是由InDel导致的,那么测序峰图就会出现套峰。
套峰产生的原因跟前面的SNP一样,具有下面几个特点:1、套峰总是两个不同类型的碱基峰套在一起;2、连续多个相同的碱基峰不会出现套峰;3、每个套峰的其中一个碱基总是在前面1个位点(缺失1个),少数在两个位点前面会找到相同的碱基峰。
如果出现了套峰,那么可以选择不重叠的前端的测序部分进行简单的物种分析,但是ITS的完整结果不能用于序列分析,需要按照第五步进行”连接载体筛选单克隆进行测序“,并且要保证能够测到两种不同序列的存在InDel变异的ITS序列。将两条序列分别命名为ITS-a和ITS-b进行后面的系统进化分析。
4、第4种情况:杂峰
如果出现多重套峰的杂峰,应该是样品污染或者测序的问题,可以重新进行ITS扩增后送测序。如果还出现这种情况,则需要将菌种进行纯化后重新扩增ITS送测序。如果反复出现这种情况,又排除了菌株污染问题,也可以按照第五步进行”连接载体筛选单克隆进行测序“,多测一些单克隆,查看一下是否存在两种以上的ITS序列。
三、ITS测序数据处理
1、如果是连接载体进行单克隆测序
建议保留ITS1和ITS4两条引物中间的所有序列,删除掉ITS1和ITS4自身的序列。
2、去掉前后测不准的区域
由于ITS序列长度只有~600 bp,因此一次测序反应就可以把全长测通,本着节约科研经费的原则,建议单端测序。
由于毛细管电泳自身的局限性,前端和后端都有测不准的区域。前端可以找一个你自己认为准确的峰作为起始碱基,宁可序列短一点,不要保留不确定的峰,因为如果因为不确定峰导致了产生多样性,这种多样性是假的。
后端的原则跟前端一样,找一个你自己认为准确的峰作为起终止碱基,宁可序列短一点,不要保留不确定的峰。
四、系统进化分析
1、将处理好的序列整理成为fasta格式的文件,放到一个文本文档中。
2、将序列进行多序列比对和系统进化分析。
如果序列数量不多,建议直接用MEGA来进行分析,具体参考:“多序列比对和系统进化分析:利用MEGA进行多序列比对和进化分析”。
如果序列数量多,可以利用服务器来进行序列分析和系统进化分析,具体可以参考:“xxxxxxxx”。
需要注意的是如果序列的来源不是单克隆测序得到的序列(扣除ITS1和ITS4序列之间的所有序列),那么由于测序质量不同会导致两端的序列的长度不同,如果不同的ITS序列之间相差显著,这种ITS长度的不同通常不会造成显著影响。也就是说,ITS长度的不同并不会影响分类学的鉴定。但是如果做同一个物种内部不同的个体之间的ITS的差异,由于不同个体的ITS本身差异很小,那么由于测序长度不同导致的差异就会影响比对结果。长度相同而且匹配的更容易聚到一起。在这种情况下,如果我们想要获得准确的结果,最好的办法就是所有的ITS序列都采用单克隆测序来做,当然由于这种办法太费时费力,可以按照下面的方法,在MEGA中完成多序列比对之后,把末端不齐的序列给删除掉。
(我们在这里不考虑全基因组重测序的分类方法,因为重测序也有自身的问题,在其他教程中讨论)
五、连接载体对单克隆进行测序(选做)
敬请期待








评论区