原创内容,欢迎转载,转载请注明出处
主笔:胡春辉
导读
- 北京时间2024年10月14日,诺贝尔奖揭晓。其中诺贝尔化学奖获奖者有三位科学家,其中两位来并非来自于传统的科研院所,而是来自于Google的DeepMind公司,两位诺贝尔奖获得者因为利用AI技术预测蛋白质结构的卓越贡献而获得了诺贝尔奖。
- 2020年11月,基于AI的蛋白质预测工具AlphaFold2发布,真正的将蛋白预测的功能提高到了几乎接近于X光衍射和冷冻电镜的水平,而且整个数据和工具是免费开源的,免费的供全人类科研圈使用。
- 由于AlphaFold2的预测速度较慢,再加上当时人工智能还没有大规模的应用,人类的显卡算力还没有像现在这么大。因此当时的AlphaFold2的在线预测非常慢,而且有时候需要排队很多天。当时我们都是拜托做蛋白结构的同学帮忙用线下的服务器进行蛋白三维结构预测。
- 在2024年5月AlphaFold3发布了,进一步提高了蛋白质三维结构预测的精确度。而且正好赶上了AI大模型爆发式增长的年代,AI算力大幅度增加。现在AlphaFold3的在线预测数量大幅度增加,每个人每天可以免费的预测20个蛋白,包括超长蛋白都可以预测。因此现在利用AlphaFold3进行蛋白结构预测,基于蛋白结构进行相关的蛋白质功能研究应该已经成为了每个研究人员都应该掌握的基础能力,就像当初的Blast等工具的使用。
- 我们在本地服务器部署了AlphaFold3,结果发现线下预测的时间需要几十分钟,跟线上比差远了,估计应该是服务器算力的问题。由于我们没有做过蛋白结构相关工作,所以得到了蛋白质三维结构之后能够做什么目前还不清楚,正在学习中。
- 除了AlphaFold3之外,还有很多其他的蛋白结构预测的工具,因为AlphaFold3的预测能力已经达到了非常高的精度,对于我们这些非蛋白结构学家而言足够用了。如果国产的蛋白预测工具有独特的功能,后面可能会添加,比如说百度的螺旋桨团队研发的HelixFold3在常规分子结构预测任务重就达到了AlphaFold3的水平。
一、最基本的蛋白质三维结构预测
(一)、AlphaFold3在线服务器预测蛋白结构
如果只有几个蛋白,直接用在线工具进行分析就行,比本地快多了。本地部署看不出来有什么优势。
1、登录
首先要打开AlphaFold3服务器,需要科学上网和Google账号。
2、输入蛋白序列开始预测
2026年开始每天免费的分析额度已经增加到了30个,应该是AI的发展使得Google的算力提升很快,有了额外的算力分给AlphaFold。
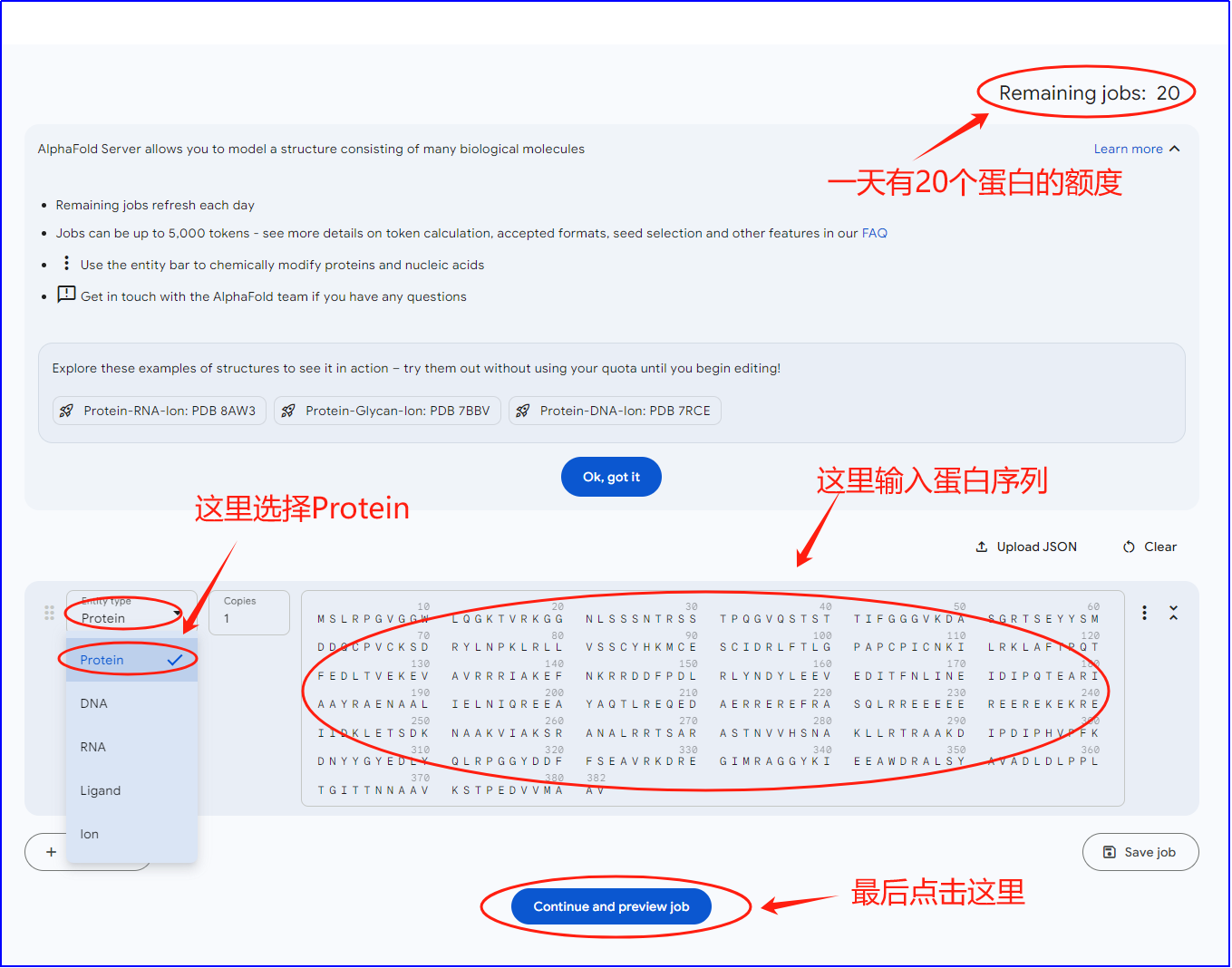
[ ]
]
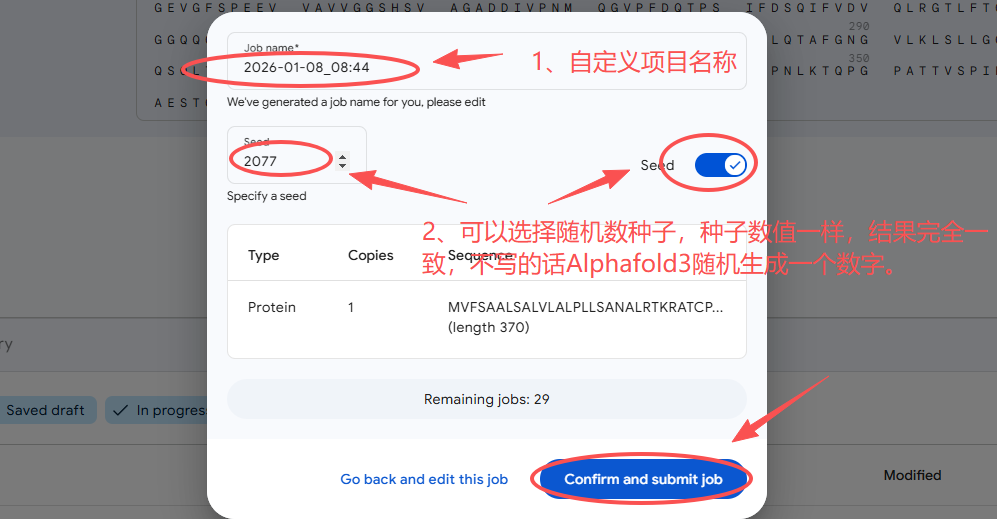
3、查看结果
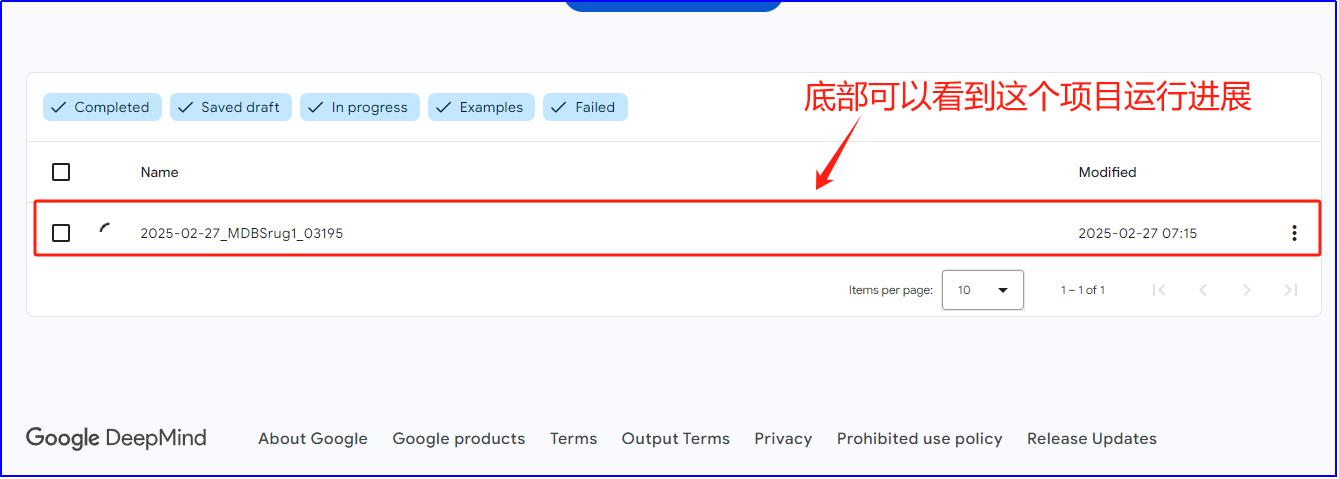
几分钟就可以预测一个蛋白的三维结构,预测完成后项目前面会显示对钩,点击就可以进入查看结果的界面。
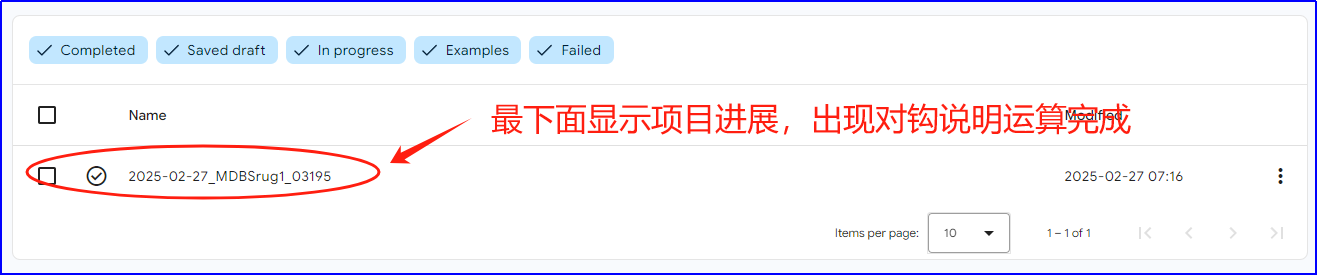
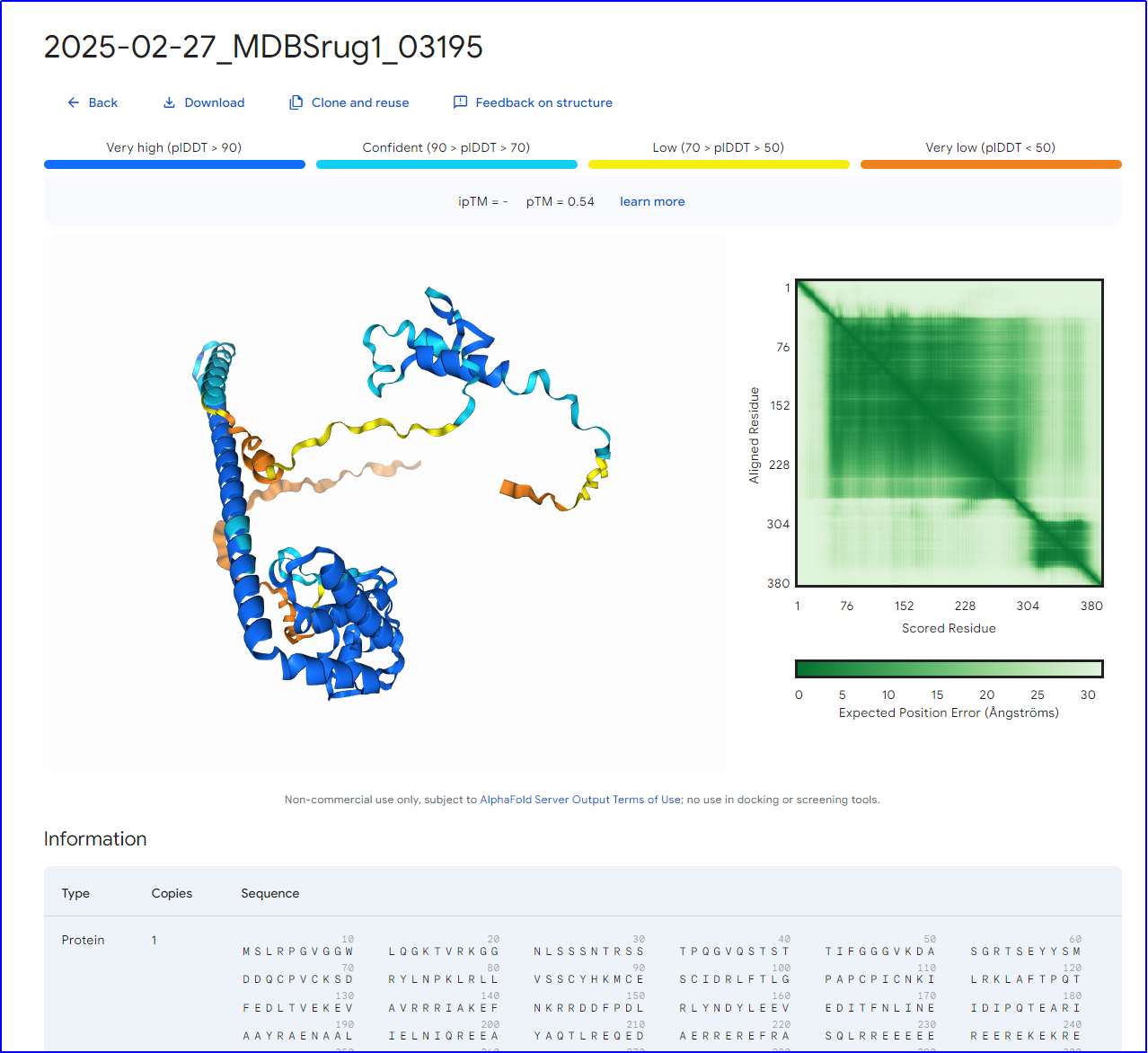
(二)、利用Singularity在本地服务器进行蛋白质三维结构预测
AlphaFold3工具本地部署主要有三种方式,AlphaFold3的GitHub官网(https://github.com/google-deepmind/alphafold3/blob/main/docs/installation.md)介绍的主要是Docker容器方法和Singularity方法进行安装使用。B站上很多博主介绍了利用Conda安装的虚拟环境进行安装。
根据我个人安装的过程,我认为Singularity容器是一种最方便简单的部署过程,下面是用本地的AlphaFold3进行蛋白结构预测的过程。
1、文件准备
如果要在本地进行AlphaFold3的蛋白质结构预测需要做下面的准备和准备一些文件。
Nvidia显卡的驱动安装和cuda驱动安装。
这部分比较简单,根据DeepSeek的提示来进行安装就行了,基本上都没有问题。

依赖数据库:600多Gb
利用官网上面提供的工具进行下载就行了,国内网络都可以下载,速度很快。
git clone https://github.com/google-deepmind/alphafold3.git cd alphafold3 chmod 777 fetch_databases.sh Sh fetch_databases.sh [<DB_DIR>]
获取模型参数:1G大小
查看上面的官网的地址(https://forms.gle/svvpY4u2jsHEwWYS6),需要注意的是应该用科研院所的邮箱地址来接收模型文件下载地址。
json格式的蛋白输入文件
下面是官网给出来的一个标准的json文件格式。
我们自己只需要准备fasta格式的文件就行,用脚本转换成为alphafold3需要的文件。
{
"name": "MDBSrug1_01943.1",
"sequences": [
{
"protein": {
"id": [
"A"
],
"sequence": "MPRPRNVVPAVHKPITEATKAFEEVQKVAQIETIINEAKEFDGKVVVVVSRT"
}
}
],
"modelSeeds": [
2025
],
"dialect": "alphafold3",
"version": 1
}
Singularity容器
2、将蛋白的fasta格式转换成为AlphaFold3使用的json格式
我自己编写了一个python脚本(下载连接)可以将fasta格式整理成为AlphaFold3的格式:
python3 af3-fasta2json.py proteins.fa
运行之后就会根据蛋白名称生成一系列的json格式文件,每个蛋白一个json格式文件。
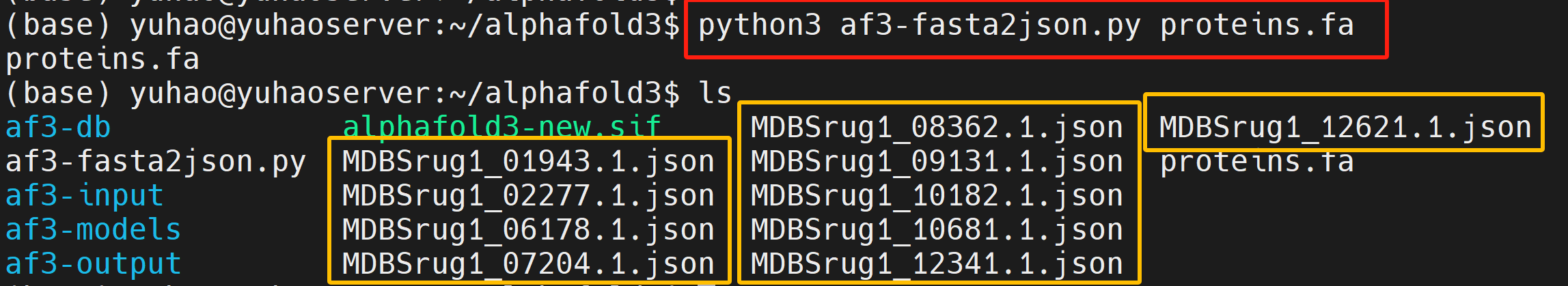
最后一步需要把这些json文件都转移到af3-input文件夹中。
mv *json af3-input
3、进行蛋白结构预测
在当前目录下运行下面的代码,如果其他目录需要修改路径。
singularity exec \
--nv \
--bind ./af3-input:/root/af_input \
--bind ./af3-output:/root/af_output \
--bind ./af3-models:/root/models \
--bind ./af3-db:/root/public_databases \
alphafold3-new.sif \
python /app/alphafold/run_alphafold.py \
--input_dir=/root/af_input \
--model_dir=/root/models \
--db_dir=/root/public_databases \
--output_dir=/root/af_output
大概几十分钟就可以预测完成,在af3-output目录下就会生成以蛋白名字作为目录名的结果文件。
由于不同的singularity容器的设置可能不一样,所以需要根据自己的容器来进行修改。
我认为Singularity使用AlphaFold3是最简单的,基本上就是傻瓜安装,失败的可能性很小,也不会影响linux系统本身,在网络连接困难的情况下可以使用本地预测的方法。Singularity的安装要比Docker容器安装简单。
有需要安装使用Singularity容器或者需要数据库文件的可以直接联系我们,你应该是能很容易找到我们的。
如果能够在线预测,建议在线预测,因为本地预测速度太慢了,在线预测多申请几个账号是最优选择。
二、更为复杂的应用:翻译后修饰和含离子、含配体的蛋白结构预测
(一)、例子
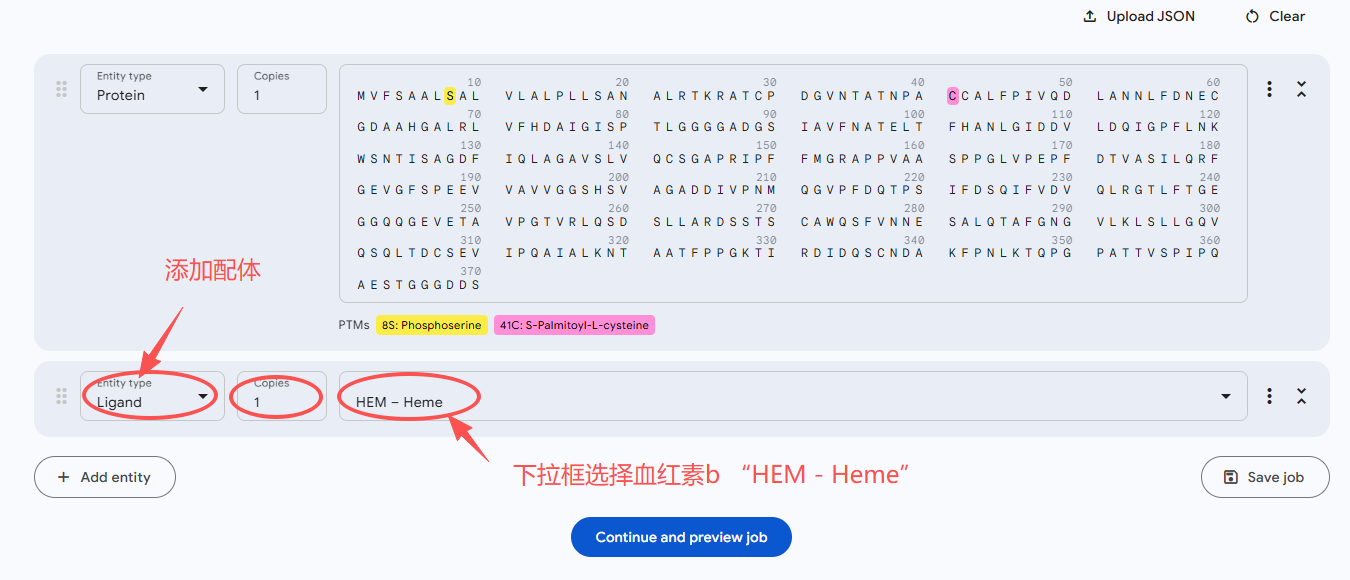
1、上面的例子只是最简单的蛋白结构预测的例子,实际上很多蛋白并不是只有氨基酸序列,而是存在很多的配体和翻译后修饰,所以这一部分就来预测一下含有配体、翻译后修饰的蛋白的结构。
2、应用的例子是一个来自于大球盖菇的锰过氧化物酶,II型的锰过氧化物酶采用血红素b(Iron Protoporphyrin IX)作为配体,此外还添加翻译后修饰的信息。
由于没有翻译后修饰的信息,所以我们这里就自己假设两个翻译后修饰,在第8位的S上面有磷酸化修饰,在第41位的C上面有棕榈酰化修饰。
3、蛋白序列如下:
>MDBSrug1_08254.1
MVFSAALSALVLALPLLSANALRTKRATCPDGVNTATNPACCALFPIVQDLANNLFDNECGDAAHGALRLVFHDAIGISPTLGGGGADGSIAVFNATELTFHANLGIDDVLDQIGPFLNKWSNTISAGDFIQLAGAVSLVQCSGAPRIPFFMGRAPPVAASPPGLVPEPFDTVASILQRFGEVGFSPEEVVAVVGGSHSVAGADDIVPNMQGVPFDQTPSIFDSQIFVDVQLRGTLFTGEGGQQGEVETAVPGTVRLQSDSLLARDSSTSCAWQSFVNNESALQTAFGNGVLKLSLLGQVQSQLTDCSEVIPQAIALKNTAATFPPGKTIRDIDQSCNDAKFPNLKTQPGPATTVSPIPQAESTGGGDDS
(二)、AlphaFold3在线服务器预测蛋白结构
如果只有几个蛋白,直接用在线工具进行分析就行,比本地快多了。本地部署在单纯的结构预测方面没有优势,优势体现在蛋白与小分子的分子对接上面(参考本网站另一个教程)。
1、登录
首先要打开AlphaFold3服务器。
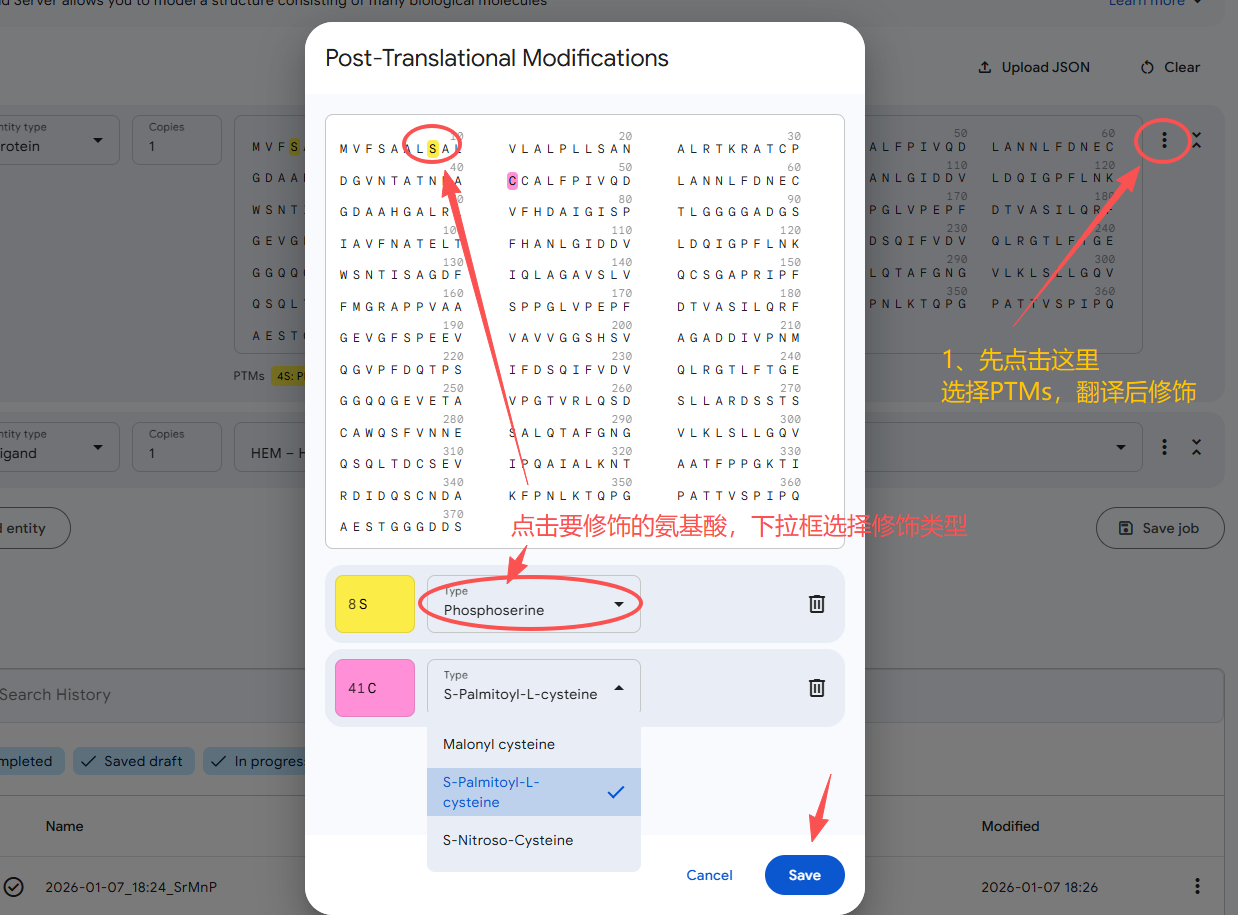
2、输入蛋白序列和翻译后修饰的具体位点和修饰类型
3、添加配体信息,配体只能从下拉框中选择,不能添加下拉框中没有的配体类型
4、蛋白信息输入之后就可以选择 “Continue and preview job” 开始进行预测了。
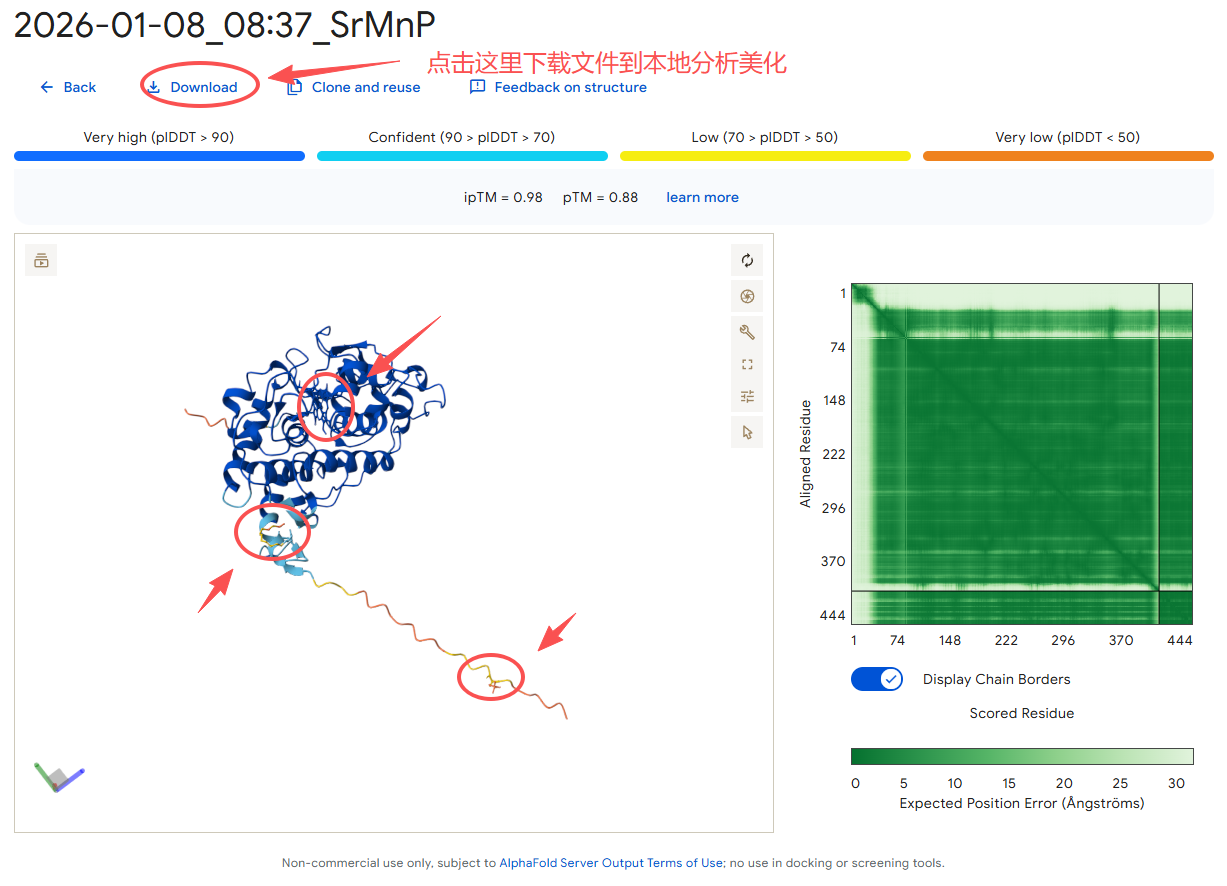
5、1-2分钟就可以完成运行,直接可以在服务器上查看结果。
可以看到蛋白结构可信度非常高,而且能够看到添加的血红素配体和2个翻译后修饰都成功的生成了。
(三)、本地服务器预测
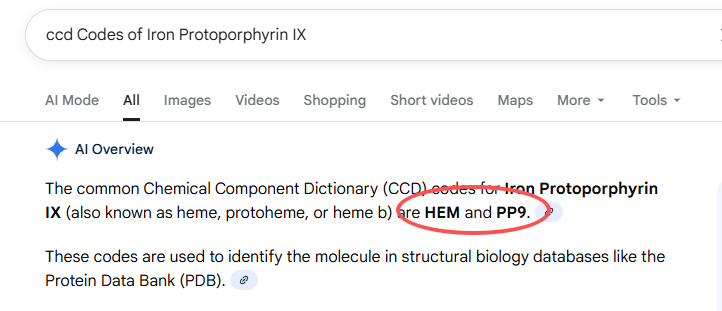
1、修饰和配体的CCD Codes
本地服务器运行首先要准确的找到要进行修饰的类型的CCD Codes是什么,以及配体的CCD Codes是什么。
这一点可以直接问AI,也可以直接到Google上面去搜索(如下图)
本案例使用的2个修饰和1个配体的CCD Codes分别如下:
| 配体/修饰 | CCD Codes |
|---|---|
| 磷酸化丝氨酸 (Phosphoserine) | SEP |
| 磷酸化苏氨酸 (Phosphothreonine) | TPO |
| 磷酸化酪氨酸 (Phosphotyrosine) | PTR |
| 磷酸化组氨酸 (Phosphohistidine) | NEP |
| S-palmitoyl-L-cysteine S-棕榈酰半胱氨酸 | P1L |
| 3-羟基脯氨酸 | HY3 |
2、根据AlphaFold3的input的规则来写json格式文件
json格式的蛋白输入文件
根据官网给出来的标准格式进行修改的,下面只是最简单的格式,详细的格式看官网的教程:AlphaFold 3 input格式
{ "name": "prostrucgly_SrMnP", "modelSeeds": [2077], "sequences": [ { "protein": { "id": "A", "sequence": "MVFSAALSALVLALPLLSANALRTKRATCPDGVNTATNPACCALFPIVQDLANNLFDNECGDAAHGALRLVFHDAIGISPTLGGGGADGSIAVFNATELTFHANLGIDDVLDQIGPFLNKWSNTISAGDFIQLAGAVSLVQCSGAPRIPFFMGRAPPVAASPPGLVPEPFDTVASILQRFGEVGFSPEEVVAVVGGSHSVAGADDIVPNMQGVPFDQTPSIFDSQIFVDVQLRGTLFTGEGGQQGEVETAVPGTVRLQSDSLLARDSSTSCAWQSFVNNESALQTAFGNGVLKLSLLGQVQSQLTDCSEVIPQAIALKNTAATFPPGKTIRDIDQSCNDAKFPNLKTQPGPATTVSPIPQAESTGGGDDS", "modifications": [ { "ptmType": "SEP", "ptmPosition": 8}, { "ptmType": "P1L", "ptmPosition": 41} ] } }, { "ligand": { "id": "H", "ccdCodes": ["HEM"] } } ], "dialect": "alphafold3", "version": 1 }
最后一步需要把json文件都转移到af3-input文件夹中(按照我们的代码是这样的,目录自己随意选择)。
mv *json af3-input
3、进行蛋白结构预测
在当前目录下运行下面的代码,如果其他目录需要修改路径。
singularity exec \
--nv \
--bind ./af3-input:/root/af_input \
--bind ./af3-output:/root/af_output \
--bind ./af3-models:/root/models \
--bind ./af3-db:/root/public_databases \
alphafold3-new.sif \
python /app/alphafold/run_alphafold.py \
--input_dir=/root/af_input \
--model_dir=/root/models \
--db_dir=/root/public_databases \
--output_dir=/root/af_output
大概而是分钟就可以预测完成,在af3-output目录下就会生成以蛋白名字作为目录名的结果文件。
4、下面是本地预测的结果,可以看出来跟在线预测的完全一样



评论区