原创内容,欢迎转载,转载请注明出处
主笔:于浩
导读
- AlphaFold3判断结构预测的结果准确性不再只看单一的分数,而是提供了一套完整的评估工具(包含局部置信度、全局拓扑及链间相互作用)帮助研究者判断结果的可靠性:
- plDDT:评估每个原子的局部可靠性(对于 PTM 和小分子同样适用)。
- pTM:聚合预测精度。
- PAE (Predicted Aligned Error):评估结构域之间或链之间的相对位置信心。
- ipTM:评估界面结合的质量
- 传统工具例如 AutoDock Vina 的打分是使用能量函数(范德华力、静电引力等)来打分,但是AlphaFold3 (AF3) 的置信度评估体系(pLDDT, PAE, pTM, ipTM)不是简单的物理能量计算,而是基于深度学习的端到端预测。
一、置信度评估
(一)、plDDT (预测局部距离差测试)
这是衡量原子/残基级别局部可靠性的核心指标。
- 定义:预测每个残基与其真实结构在 原子位置上的重合程度。
- 范围:0 - 100
- 区间说明:
- > 90:极高置信度:结构细节(甚至侧链取向)非常可靠,可用于药物设计(比如活性中心位置的plDDT都大于90,则分子对接结果可信度高)。
- 70 - 90:高置信度:二级结构和骨架拓扑可靠。
- 50 - 70:低置信度:结果仅供参考,可能属于柔性区域。
- < 50:极低置信度(非结构化):通常预示该区域是固有无序区 (IDR),在生理条件下没有固定形状。
(二)、pTM (聚合预测精度)
pTM 是 AlphaFold 系列模型中用于评估全局拓扑结构(Global Fold)准确性的核心参数。它的设计灵感来源于结构生物学中经典的 TM-score。
- 范围:0.0 - 1.0,分值越高,说明模型对其预测的整体形状越有信心。
- 区间说明:类似于传统的 TM-score。若 > 0.5,说明预测的全局折叠拓扑大致正确。
| pTM 值区间 | 预测质量评价 | 生物学含义与应用建议 |
|---|---|---|
| > 0.8 | 极高精度 | 全局折叠非常可靠。 蛋白质的拓扑结构、核心区域的堆积基本完全正确,可用于高质量的功能机制分析。 |
| 0.5 - 0.8 | 中等精度 | 拓扑结构大致正确。 蛋白质的主体折叠(如螺旋和折叠的排列)是可靠的,但结构域之间的相对位置可能存在小幅偏差。 |
| 0.2 - 0.5 | 低精度 | 不可信的折叠。 结构可能只有局部小片段(如单个 alpha-螺旋)是正确的,但整体像“乱麻”一样。通常见于极度缺乏同源序列的蛋白。 |
| < 0.2 | 无序/随机 | 完全不可信。 序列可能是固有无序蛋白 (IDP),在生理状态下可能根本没有固定的三维结构。 |
(三)、ipTM (界面聚合预测精度,chain pair ipTM)
1、ipTM 是 AlphaFold3 (AF3) 中最关键的参数之一,专门用于衡量不同分子链之间(如蛋白与蛋白、蛋白与小分子、蛋白与核酸)结合界面的预测准确性。如果说 pTM 是看“单体折叠得像不像”,那么 ipTM 就是看“它们凑在一起凑得对不对”。
2、ipTM 的全称是“界面预测模板建模分数”。它专注于评估复合物的相对位置。 在结构生物学中,即使两个蛋白质各自的形状都预测对了,如果它们相互结合的角度或位置错了,整个复合物的功能模型就是错误的。ipTM 正是为了捕捉这种“界面精度”而设计的。
- 范围:0.0 - 1.0
- 区间说明:
| ipTM 值区间 | 预测质量评价 | 生物学含义与应用建议 |
|---|---|---|
| > 0.8 | 极高置信度 | 界面非常准确。 氨基酸侧链与底物/另一条链的接触细节高度可信,可直接用于药物筛选或催化机制研究。 |
| 0.6 - 0.8 | 高置信度 | 结合模式基本正确。 虽然具体的氢键或范德华力细节可能有微调,但整体的对接位置和朝向是可靠的。 |
| 0.4 - 0.6 | 中低置信度 | “灰色地带”。 模型认为它们可能结合,但界面可能不稳定或存在多个可能的姿态。建议结合实验数据或 PAE 图进一步验证。 |
| < 0.4 | 不可信/未结合 | 对接失败。 这种分值通常意味着模型只是随便把两个分子摆在了一起,它们之间没有真实的相互作用,或者该蛋白根本不结合这个分子。 |
(四)、PAE(预测对齐误差)
1、PAE 是 AlphaFold 系列中用于评估**结构域之间(Domain-to-Domain)或链之间(Chain-to-Chain)**相对位置信心的关键指标。它是判断蛋白质“整体构象稳定性”最重要的工具。
2、PAE 代表“预测对齐误差”。它衡量的是:如果我们将预测结构与真实结构在残基 i上进行对齐,那么残基 j 的预测位置与真实位置之间的距离误差是多少?
- 直观理解:它告诉我们模型对于“残基 i 和残基 j 之间的相对位置”有多大的把握。
- 不对称性:虽然在大多数情况下它是近似对称的,但理论上残基 i 对 j 的误差预测与 j 对 i 的可能略有不同。
3、范围
PAE 的单位是 埃 (Å)。数值越小,表示位置越确定。
| PAE 值 (Å) | 视觉颜色(热图) | 生物学含义 |
|---|---|---|
| 0 - 5 | 深绿色 | 极高信心。两个残基(或结构域)之间的相对位置非常固定且准确,通常属于同一个坚固的折叠单元内部。 |
| 5 - 12 | 浅绿色 | 高信心。结构域之间的相对排布大致可靠,可能存在轻微的柔性摆动。 |
| 12 - 20 | 浅绿/黄色 | 低信心。模型认为这两个部分可能在一起,但它们之间的取向是不确定的,可能存在较大的构象变化。 |
| > 25 / 封顶 | 红色/白色 | 无信心。这两个残基之间没有固定的相对位置。通常意味着它们被长而柔软的 Linker(连接子) 隔开,或者是完全独立 |
(五)、Ranking Score (排名分数)
在下载到本地的结果中( “summary_confidences.json” 文件),会有一个 ranking_score,它是通过以下公式综合了 pTM 和 ipTM 的结果:
Ranking Score = 0.8×ipTM + 0.2×pTM + 其他界面分数
为什么要这么算? 因为在复合物预测中,界面的准确性(ipTM)重要性远高于单个蛋白的折叠(pTM)。即使蛋白折叠只有 0.5 分,但如果界面能达到 0.8 分,这个复合物模型依然具有极高的研究价值。
二、如何综合性评估结果
(一)、快速判断结果是否准确
先看 pLDDT 和 PTM:确定蛋白自己有没有“散架”,关注蛋白内部的结构是否准确。
再看 PAE 热图:确定复合物内部的“块”之间是否有深绿连接。如果没有,即使 pLDDT 再高,它们也不是一个稳定的整体。
最后看 ipTM:给结合强度打分。如果 ,无论 3D 图看起来多漂亮,都不要轻易相信那个结合姿态。
| 你的问题 | 查看参数 | 理想阈值 |
|---|---|---|
| 某个氨基酸位置对不对? | plDDT | > 70 |
| 全局拓扑结构(蛋白折叠)对不对? | PTM | > 0.6 |
| 两个结构域的相对位置对吗? | PAE | < 5 Å (深绿) |
| 蛋白和 DNA 真的结合吗? | ipTM | > 0.6 |
| 小分子(配体)位置准吗? | ipTM | > 0.5 |
(二)、一些典型结果
1、带柔性连接子的双结构域蛋白
参数数值:
- pLDDT: 结构域内部 > 85,但 Linker 区域 < 50(橙红色)。
- pTM: 0.65(中等)。
- ipTM: 0.30(极低)。
- PAE: PAE 图中出现两个明显的深绿色对角线方块,但方块之间是纯白色。
综合结论:结构域内部准确,但相对位置是随机的。
含义:两个蛋白自己折叠得很稳,但它们之间并没有相互作用。此时不能根据预测图去猜测 A 和 B 之间如何相互作用。
2、酶与完全不匹配的小分子强制对接
参数数值:
- pLDDT: 蛋白部分 88(很好),但小分子部分仅 40(红色)。
- pTM: 0.85(蛋白折叠没问题)。
- ipTM: 0.25(极低)。
- PAE: 蛋白内部深绿,但蛋白与小分子的交界处是一片浅色。
综合结论:蛋白折叠(结构)是对的,但对接是“幻觉”。
含义:模型为了完成任务,勉强把小分子放在了表面。但由于 ipTM 太低且 PAE 太大,这个小分子的位置没有任何参考价值,它可能只是“滑”过了蛋白表面,并没有结合。
三、从哪里查看评估结果
(一)、AlphaFold3在线置信度结果
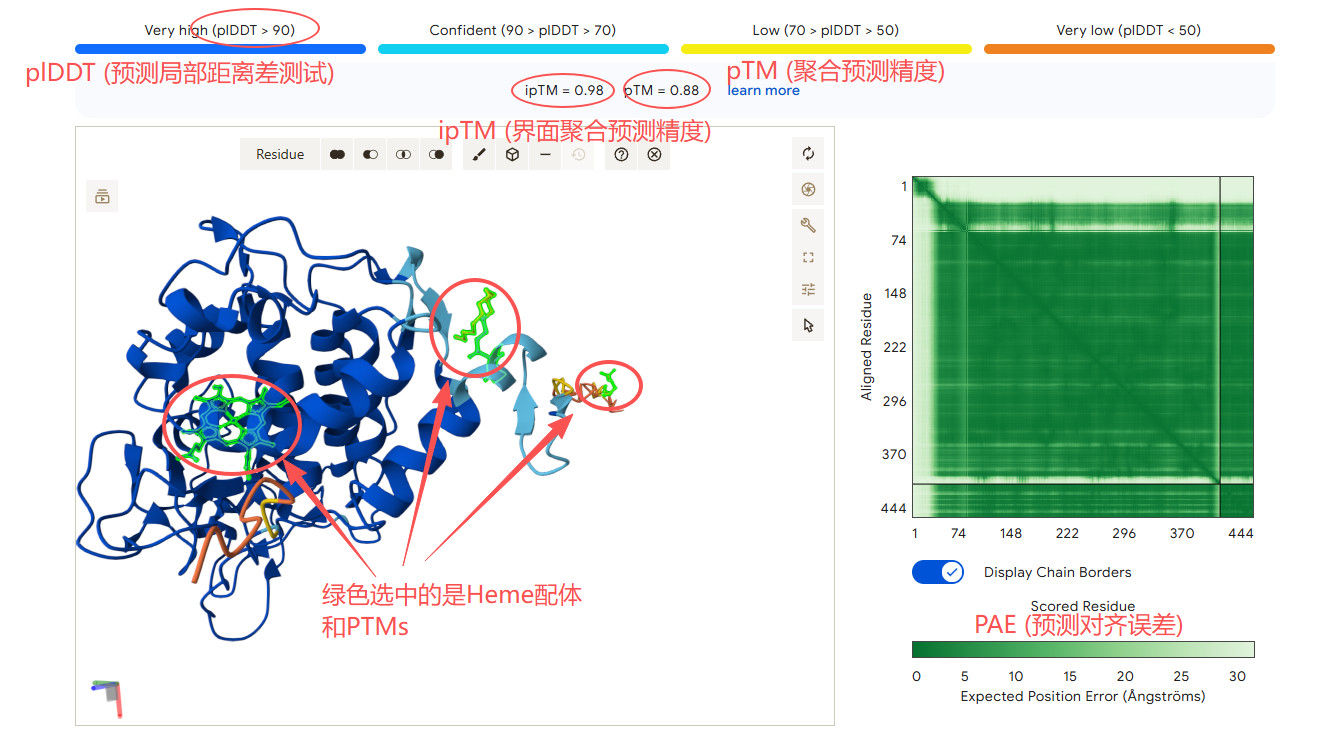
打开在线服务器预测的结果,就可以看到四个主要的置信度评估参数,具体的位置参考下图。
这个例子还是之前在蛋白结构预测的教程里面的结果。
(二)、AlphaFold3置信度结果本地查看
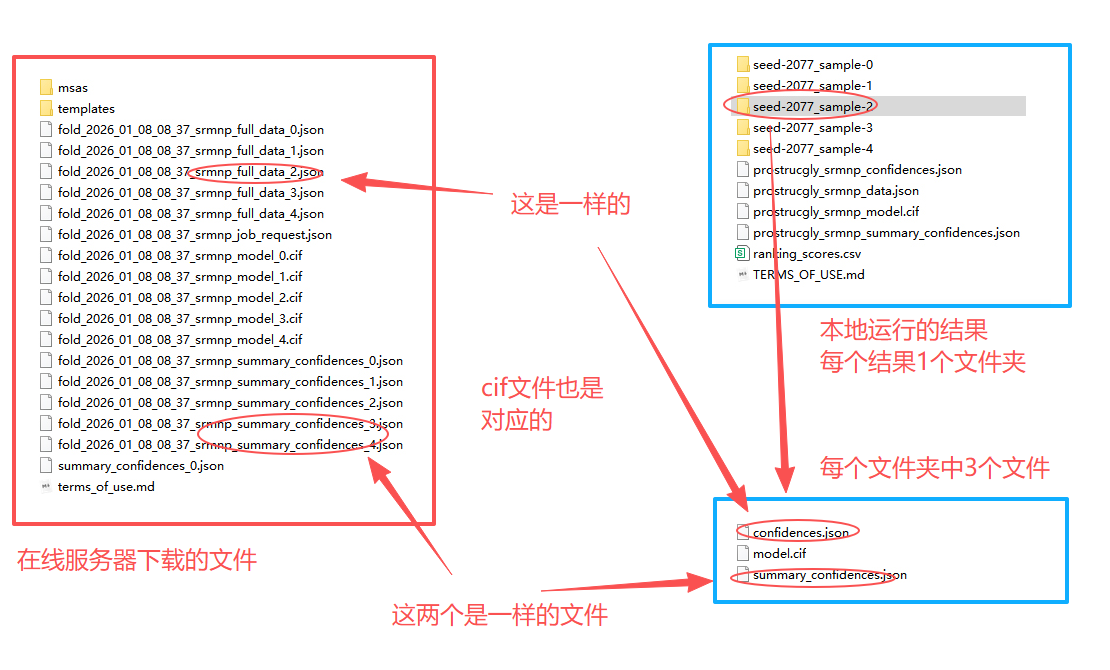
1、预测的结果不仅可以在线查看而且可以把结果保存到本地再进行查看。
2、在线下载的结果和本地Linux服务器运行的结果在文件命名和文件目录存在差异,具体的查看下图。
3、下面以本地运行结果的文件名字来查看一下上面的四个参数从哪里查看。
(1)从 “summary_confidences.json” 中可以查看 ipTM 和 PTM 值。
{
"chain_iptm": [
0.98,
0.98
],
"chain_pair_iptm": [
[
0.87,
0.98
],
[
0.98,
0.84
]
],
"chain_pair_pae_min": [
[
0.76,
0.82
],
[
0.94,
0.76
]
],
"chain_ptm": [
0.87,
0.84
],
"fraction_disordered": 0.09,
"has_clash": 0.0,
"iptm": 0.98, # iptm值在这里
"ptm": 0.89, # ptm值在这里
"ranking_score": 1.0 # ranking_score值在这里
}
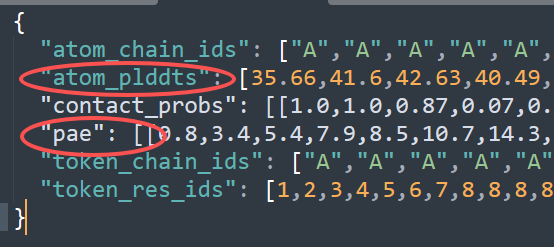
(2)从 “” 文件中可以查看每个原子的 plDDT 值,以及查看 相对位置的 PAE (预测对齐误差) 值。


评论区