原创内容,欢迎转载,转载请注明出处
主笔:于浩
导读
1、本教程是AlphaFold3系列的第3篇教程,前面两个教程分别介绍了 “AlphaFold3的本地化安装” 、 “AlphaFold3预测蛋白结构” 、 “AlphaFold3预测蛋白与蛋白相互作用” 、 “蛋白与核酸相互作用“,上面的教程基本上覆盖了AlphaFold3的主要功能,本教程补上最后一个功能 ”蛋白与小分子的分子对接(相互作用)“。由于”抗体-抗原复合物结构“等研究我们不涉及所以其他复杂功能暂时就不介绍了。此外,对于翻译后修饰(PTMs)、DNA碱基修改等内容会逐渐的补充到蛋白结构预测和蛋白-DNA相互作用等教程中,也不再单独列出来了。
其他两个教程的超链接:
2、蛋白与小分子的分子对接使用最多的工具是AutoDock Vina,这个在后面还会单独做教程,到时候会简单的介绍一下蛋白小分子分子对接的一些背景,这里也不做介绍了。
注意:使用Alphafold3预测蛋白小分子相互作用不是传统的 “分子对接(Docking)” 而是属于 “联合结构预测” 的逻辑。
3、对于结果的分析后面会陆续添加。
利用AlphaFold3进行“蛋白-小分子”分子对接
(一)、本教程使用的例子
漆酶是食用菌降解木质素的主要胞外酶,漆酶除了能够作用于木质素,还能够对木质素降解产物进行分解,尤其是其中的酚类化合物。对羟基苯甲酸是木质素降解产物之一,本教程就来研究一下漆酶与对羟基苯甲酸之间的分子对接过程。
选择的漆酶是来自我们已经发表的一篇文章:
SrLac9蛋白序列如下:
>MDBSrug1_07931.1
MLSLLAVALLPAAYAAIGPRANVFIQNKIISPDGFNRSAVLAGGVQGKPTFPGPLITGEKGDTFSLNVVNELNDTTMLTSTSIHWHGLFQRGTAWADGPVGVTQCPIIPGNSFNYQFQALDQAGTYWYHSHYSTQYCDGLRGAMVIYDPNDPYKNQYDYDDDSTVITLADWYHTVAPVLGASKTPPKSDATLINGLGRFSANDTSPLAVISVVPNKRYRFRLVSISCDPNFTFSIDNHTMTIIEVDGNNVQPLSVDSIQIYAGQRYSFILQANQRPSNYWARALPNIGPAGFTGGVNSAILRYVGAPEADPTTPLVPSTRPMLETQLQPLTNPAAPGPAVPPALSNGEVIPLSFNISFNGSDFAVNNFTFQPPPMPVLLQILSGKHKAQELLPVGSVYTLPPNKVIEITIPGGTLGAPHPIHLHGHAFSVIRSAGSNVTNYNNPVRRDVVNSGSSTSDLVTIRFETDNAGPWIMHCHIDWHLDKGLAIVLAENVDGIAQLNPPVAWDQLCPAFDALPPQTFNN
(二)、AlphaFold3在线服务器进行 蛋白-小分子 互作研究
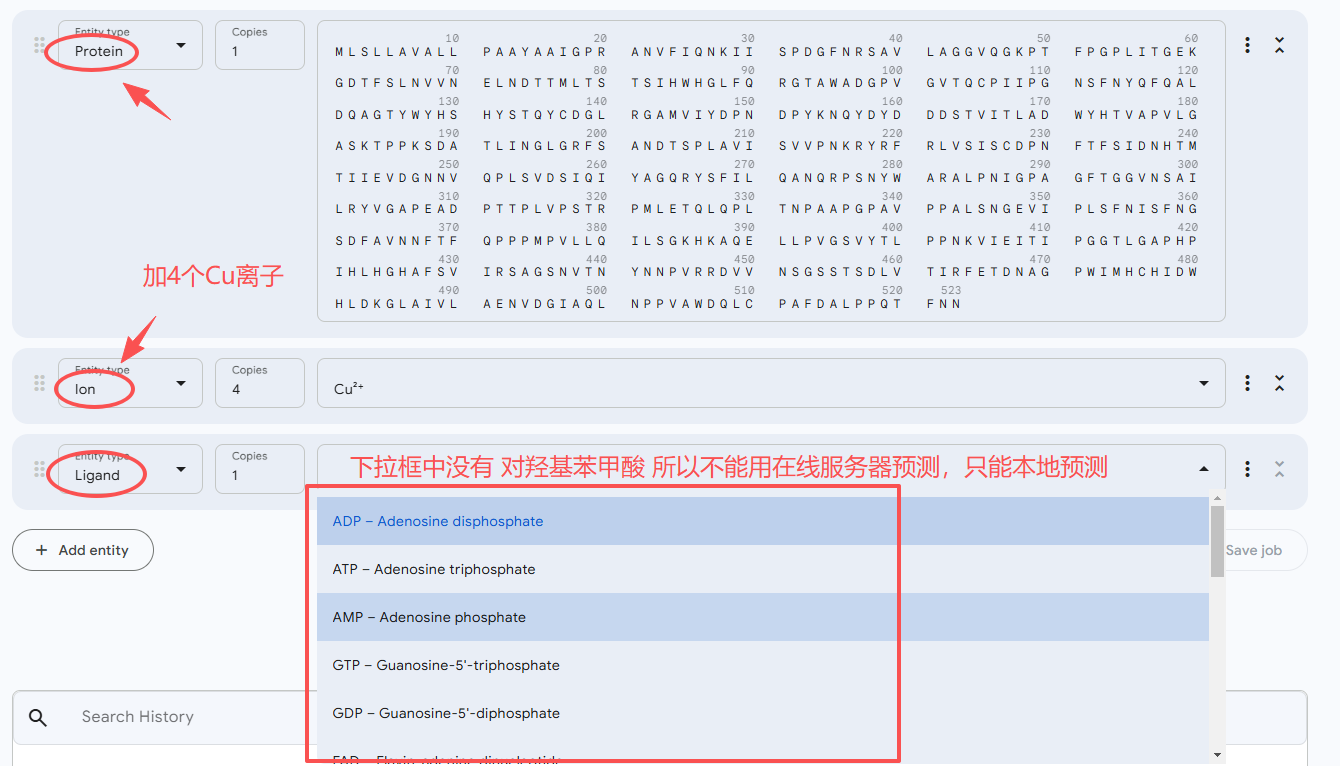
首先登录在线服务器,依次将漆酶的蛋白序列、4个Cu2+离子输入到对话框中。
小分子需要以配体(ligand)的形式添加,但是在线服务器的ligand选项中不能自己手动输入小分子的结构,因此必须要从下拉框中选择。但是下拉框中没有“对羟基苯甲酸”,因此无法用在线服务器完成本任务,只能用本地服务器。
(三)、在本地服务器进行“蛋白-小分子” 互作研究
1、小分子的结构分子准备
AlphaFold3预测蛋白与小分子相互作用时小分子需要以配体(ligand)的形式添加,配体分子的结构可以用该分子的CCD code 或者 SMILES(Simplified Molecular Input Line Entry System)表示法来表示。
2、分子的SMILES表示法查询
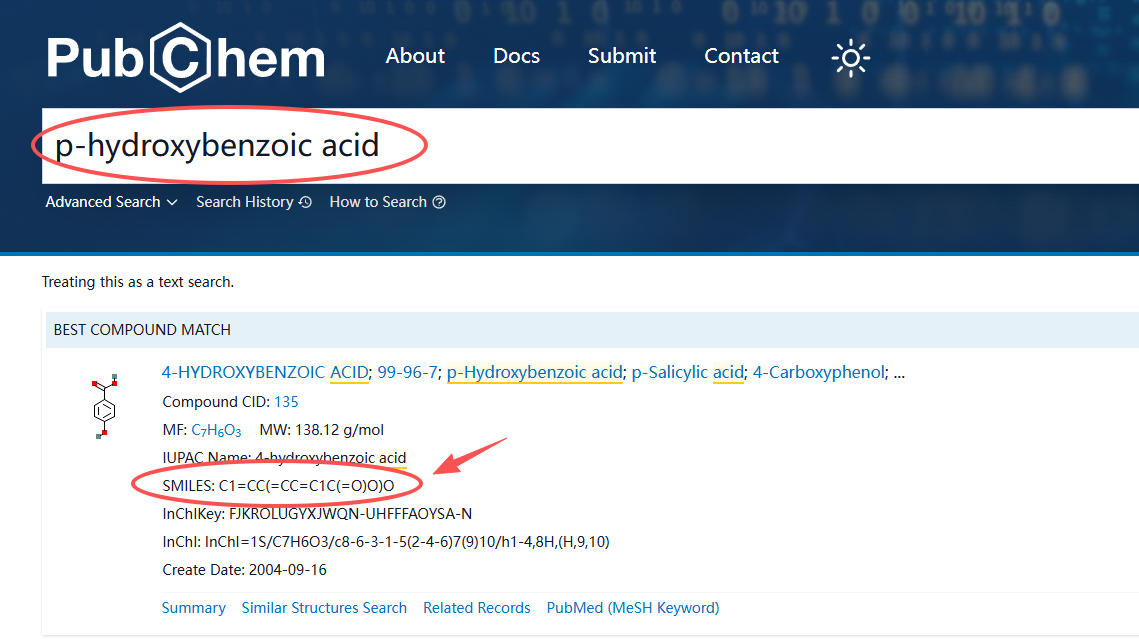
🔗 访问网址: https://pubchem.ncbi.nlm.nih.gov/
搜索 “p-hydroxybenzoic acid” ,查看 SMILES信息,可以通过CAS号来确认是否是自己想要检索的分子。
可以看到对羟基苯甲酸的SMILES表示法为:
SMILES: C1=CC(=CC=C1C(=O)O)O
3、分子的CCD Codes查询
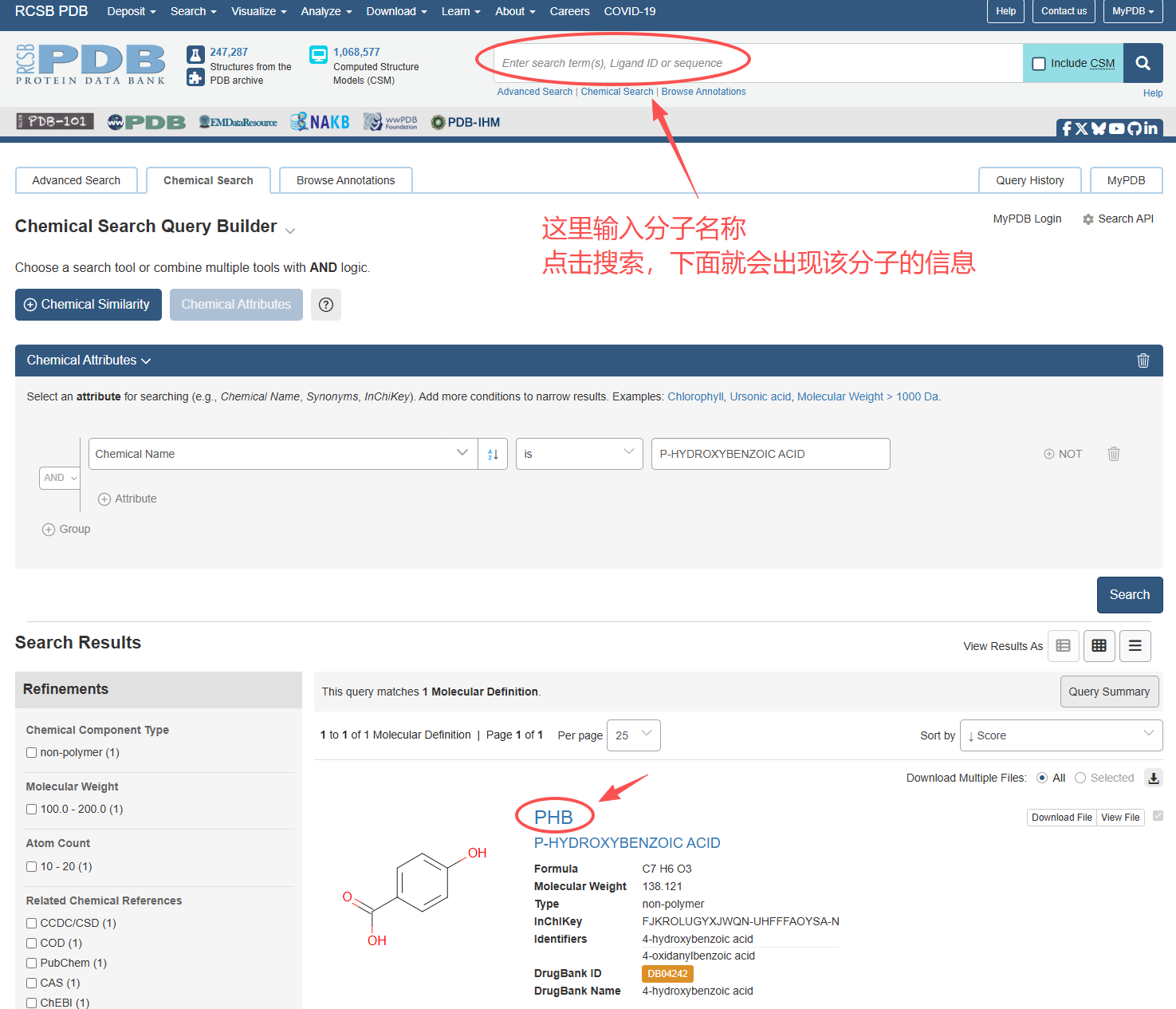
通过 RCSB PDB 官网搜索(推荐)
进入网址,在顶部搜索框输入 分子名称(如 “P-HYDROXYBENZOIC ACID”)点击进入后,URL 或页面会显示其 CCD ID(即 3 字符代码)
可以看到对羟基苯甲酸的CCD Codes为:PHB
4、Json文件准备
这里就采用SMILES的表示法来写 JSON 文件,也可以使用 CCD Codes 表示法,CCD Codes可以参考上一个教程。
Cu离子一共是4个也需要以Ligand的形式添加。
json格式的蛋白输入文件
{ "name": "promol_lacphb", "modelSeeds": [2077], "sequences": [ { "protein": { "id": "A", "sequence": "MLSLLAVALLPAAYAAIGPRANVFIQNKIISPDGFNRSAVLAGGVQGKPTFPGPLITGEKGDTFSLNVVNELNDTTMLTSTSIHWHGLFQRGTAWADGPVGVTQCPIIPGNSFNYQFQALDQAGTYWYHSHYSTQYCDGLRGAMVIYDPNDPYKNQYDYDDDSTVITLADWYHTVAPVLGASKTPPKSDATLINGLGRFSANDTSPLAVISVVPNKRYRFRLVSISCDPNFTFSIDNHTMTIIEVDGNNVQPLSVDSIQIYAGQRYSFILQANQRPSNYWARALPNIGPAGFTGGVNSAILRYVGAPEADPTTPLVPSTRPMLETQLQPLTNPAAPGPAVPPALSNGEVIPLSFNISFNGSDFAVNNFTFQPPPMPVLLQILSGKHKAQELLPVGSVYTLPPNKVIEITIPGGTLGAPHPIHLHGHAFSVIRSAGSNVTNYNNPVRRDVVNSGSSTSDLVTIRFETDNAGPWIMHCHIDWHLDKGLAIVLAENVDGIAQLNPPVAWDQLCPAFDALPPQTFNN" } }, { "ligand": { "id": ["W", "Z", "Y", "X"], "ccdCodes": ["CU"] } }, { "ligand": { "id": ["F"], "smiles": "C1=CC(=CC=C1C(=O)O)O" } } ], "dialect": "alphafold3", "version": 1 }
5、进行蛋白结构预测
json格式的文件放到 af-input 目录下面,运行代码参考之前的教程,都是一样的。
运行比较长的时间(跟服务器性能有关系),然后就会出结果,将结果下载下来,利用ChimeraX或者PyMOL打开就可以看到了。
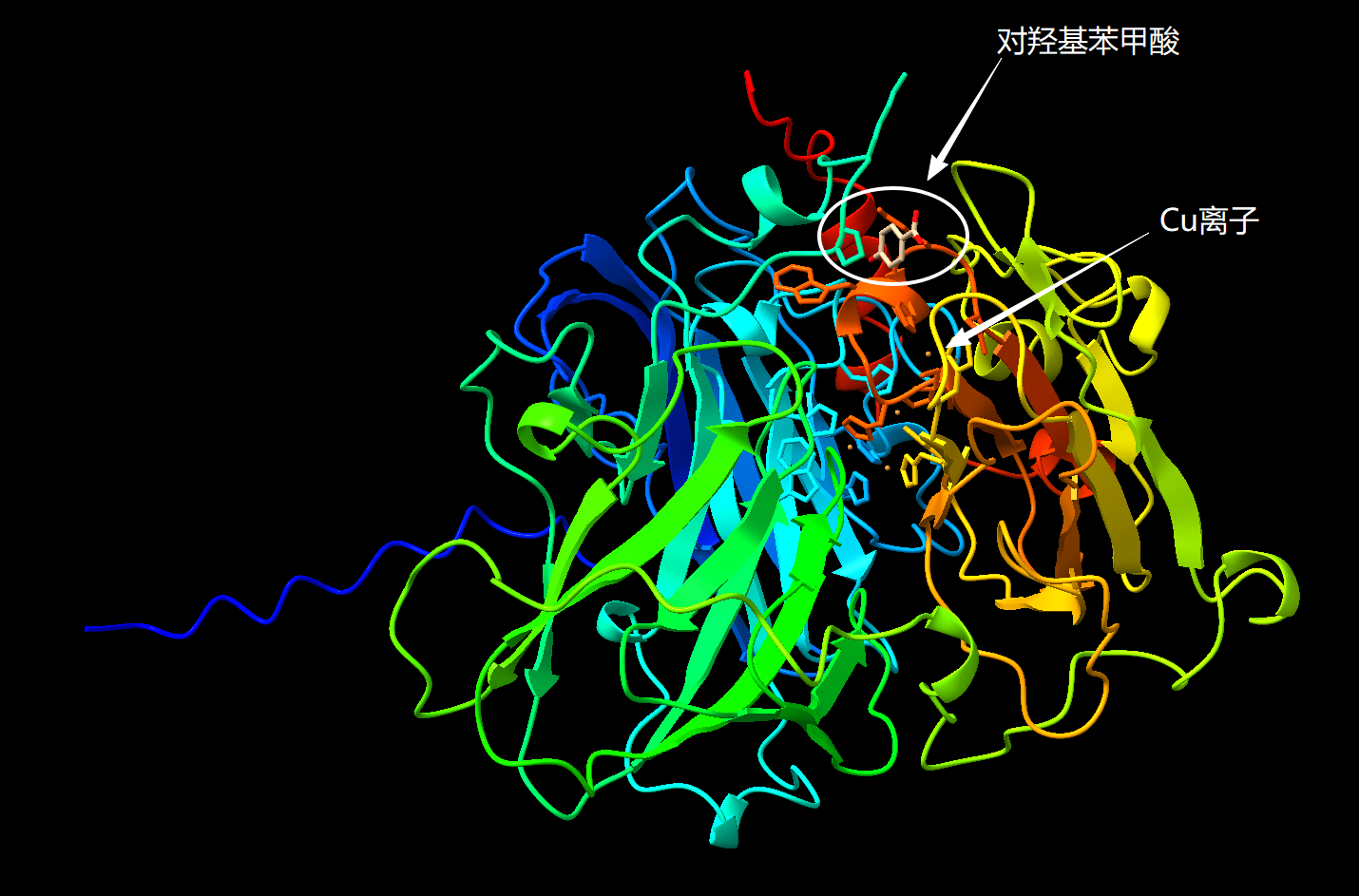
(四)、“分子对接”结果展示
下面是利用ChimeraX打开的分子对接的结果



评论区