原创内容,欢迎转载,转载请注明出处
主笔:于浩
导读
1、什么是分子对接
分子对接(Molecular Docking) 是一种计算模拟方法,用于预测小分子(如药物候选物,称为“配体”)与生物大分子(如蛋白质,称为“受体”)之间如何结合,包括:
- 它们在空间中的最佳结合构象(即配体在蛋白口袋中的位置和朝向)
- 它们的结合亲和力(通常用结合自由能 ΔG 表示,单位为 kcal/mol)
在现代结构生物学与计算机辅助药物设计等领域的发展,分子对接(Molecular Docking)已成为探索生物大分子受体与小分子配体之间相互作用模式的核心技术手段。分子对接的本质是通过计算模拟,预测小分子在蛋白质结合口袋中的最优结合姿态(Binding Pose)并估算其结合亲和力(Binding Affinity) 。随着结构生物学数据的爆炸式增长,利用高性能计算筛选潜在的先导化合物已成为药物研发的标准流程 。
2、分子对接的分类
(1)根据对分子柔性(flexibility) 的处理方式,分子对接主要分为三类:
对接类型 特点 刚性对接 蛋白和配体的构象均固定不变,仅搜索空间位置和取向。计算快,但精度低。 半柔性对接 配体构象可变(如旋转单键),蛋白骨架固定。兼顾效率与准确性,最常用。(代表性软件就是AutoDock Vina) 柔性对接 蛋白和/或配体的构象均可自由变化(如侧链柔性)。精度高,但计算量极大,耗时长。 实际应用中,半柔性对接是主流,因为大多数药物靶点结构来自X射线晶体(构象较固定),而小分子柔性可通过构象采样有效处理。
(2)根据物理和数学模型的不同,主要可以分为以下几种:
原理类型 特点 基于物理力场的原理(Classic Physics-based) 通过计算范德华力、静电引力、氢键、脱溶剂效应等能量项之和来评估结合强度。这是传统对接软件的基石。 基于几何匹配的原理(Shape Complementarity) 主要看“钥匙”和“锁”的形状是否契合,通过表面形状的互补性来快速筛选结合位点。 基于知识/经验的原理(Knowledge-based) 通过统计已知的蛋白质-配体晶体结构数据库,计算原子对之间出现的频率,转化为统计势能,频率越高认为越稳定。 基于深度学习/生成式的原理(AI-based/Generative) 不显式计算物理能量,而是通过神经网络学习海量结构数据中的空间模式,直接“生成”或“预测”最可能的坐标。
3、为什么要选择AutoDock Vina
在众多分子对接工具中,AutoDock Vina凭借其卓越的搜索效率和预测准确性,自2010年发布以来便成为该领域SCI引用率最高的开源软件 。AutoDock Vina是AutoDock Suite套件中的核心引擎,在处理大规模虚拟筛选时速度是AutoDock4高两个数量级,在评分函数的稳健性和构象搜索的广度上也实现了超越 。
选择AutoDock Vina的核心理由就是这个工具是在SCI文章里面使用最广泛的免费开源分子对接工具。我没有做过小分子开发,我也没有做过药物开发。我利用分子对接就是辅助验证我做实验的到的结论(所以做和不做不影响实验结论),所以我就要使用一个学术上最认可的工具,以增加实验结果的可信度。
假设我现在是药物开发的人员,实验是围绕分子对接结果开展的,我肯定首选商业化的软件,而且去公司参加培训,严格按照商业版软件的操作流程来进行试验。
4、AlphaFold3的分子对接
采用生成式人工智能的原理,构建最稳定的分子和蛋白复合物结构。具体的操作方式参考本网站的下面的这个教程:
一、AutoDock Vina分子对接
1.1 分子对接基本流程
\流程图.jpg)
1.1 蛋白和配体分子准备
1、蛋白3D结构文件获取
- 如果研究的蛋白已经有了晶体结构,那么直接从PDB数据库获得蛋白晶体结构文件(.pdb格式)就行,一定要选择高精度的晶体结构蛋白。
- 如果研究的蛋白没有晶体蛋白结构,那么可以使用AlphaFold3等工具预测蛋白结构用于分子对接。AlphaFold3预测蛋白结构可以参考本网站教程:AlphaFold3:利用AlphaFold3进行蛋白质三级结构预测&AlphaFold3的本地安装
AlphaFold3 生成的蛋白3D结构是 cif 格式的文件,需要转换成为 pdb 格式的文件才行。可以用 ChimeraX 打开cif格式的文件,点击 “File / Save” 选择保存成为 pdb(默认格式)文件就行了。
2、小分子2D结构文件获取
- **要求:**分子结构完整(无缺失原子),避免带电基团不合理(如游离羧基、氨基需符合生理 pH 条件)
(1)配体分子来源
已知分子:
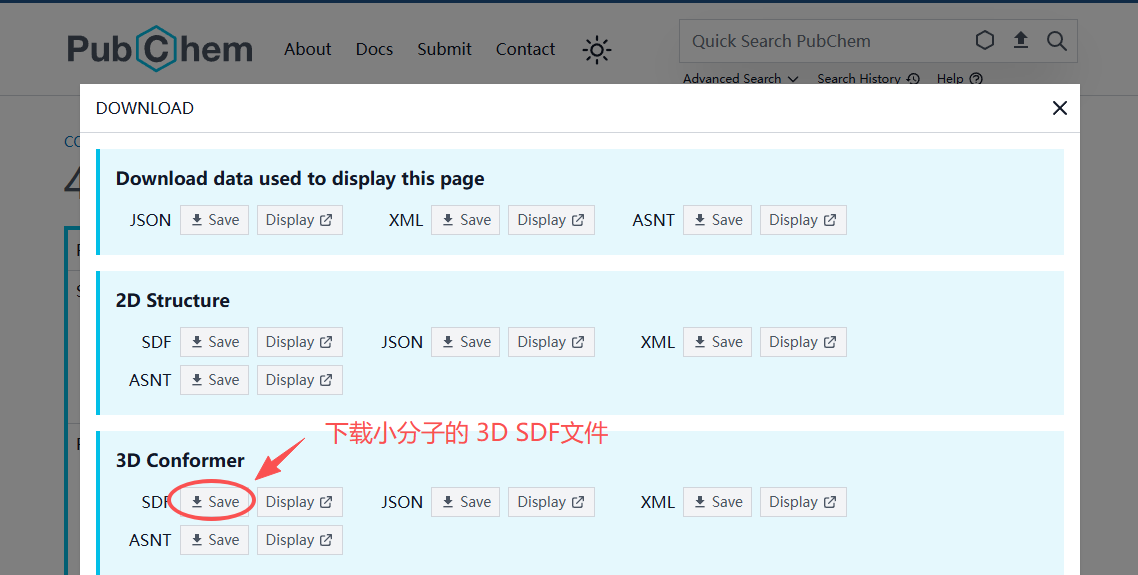
通用化合物库PubChem,含大量天然产物:🔗https://pubchem.ncbi.nlm.nih.gov/ (直接下载3D的SDF 格式)
中药数据库TCMID:🔗http://www.megabionet.org/tcmid/
含靶点活性数据的药物化学数据库ChEMBL:🔗https://www.ebi.ac.uk/chembl/
天然产物图谱数据库NPAtlas(The Natural Products Atlas):🔗 https://www.npatlas.org
Zinc数据库:🔗 https://zinc.docking.org
自主设计分子:
可以用用免费开源的 BKChem 软件生成 cml 格式文件,也可以用 ChemDraw 绘制后导出 SDF 格式文件。
也可以用PubChem的免费在线工具:https://pubchem.ncbi.nlm.nih.gov/edit3/index.html 绘制,再由其他的软件转换成为2D格式的分子文件。
3、小分子2D结构转化成为3D的结构
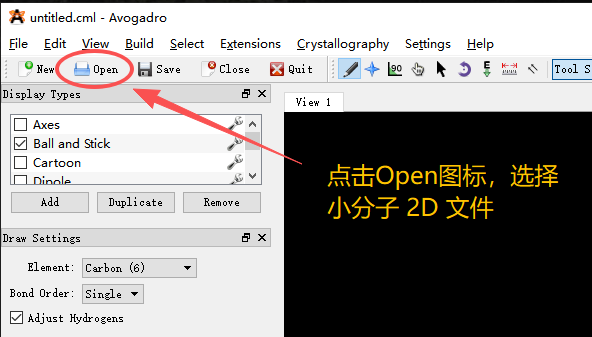
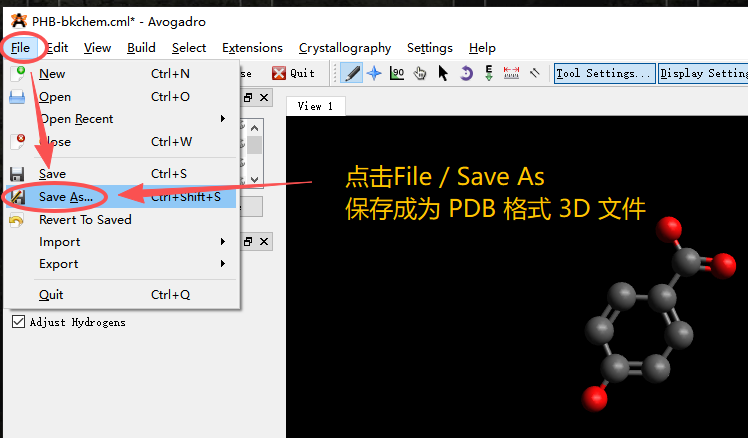
(1)小分子2D文件生成3D文件可以使用Open Babel,也可以使用图形界面 Avogadro,一个合理的初始构象能显著降低Vina在搜索过程中的内能计算偏差 。前者适合Linux服务器批量转换,后者适合在Windows中单个操作。下面介绍一个 Avogadro 格式转化过程。
初始的文件是利用 BKChem 生成的对羟基苯甲酸的 CML 格式文件。
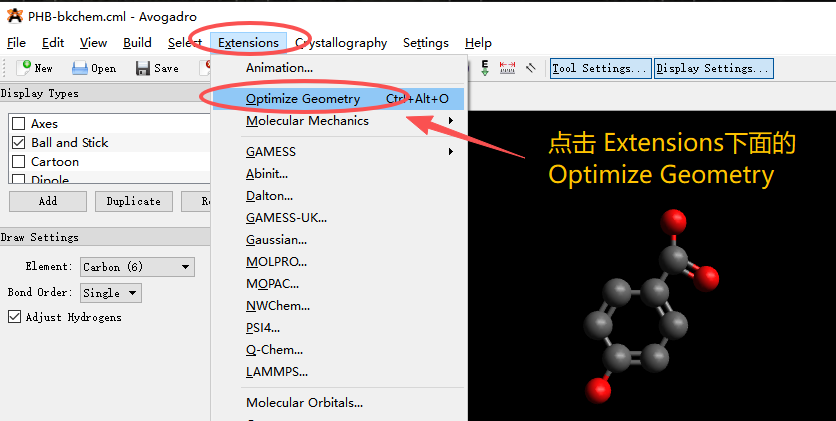
下面这一步就是用来优化立场的,也就是优化构象,因为2D的分子不能正确的体现分子在三维空间中的结构,有可能出现分子内冲突(如原子重叠),所以这步必须要优化。
1.2 蛋白预处理
1、分子对接前,必须将蛋白质受体和小分子配体转换为AutoDock Vina支持的 PDBQT 格式,这是确保对接计算准确性的关键步骤,ADT可以完成该步骤。
不需要为蛋白和小分子来分配电荷,因为AutoDock Vina内部会自动处理电荷,不依赖Gasteiger电荷,在运行的时候会根据原子元素、键连环境推断部分电荷和原子类型,因此会自动忽略输入文件中的电荷。但是需要为小分子和蛋白加氢,因为氢键直接参与关键相互作用的识别与打分。
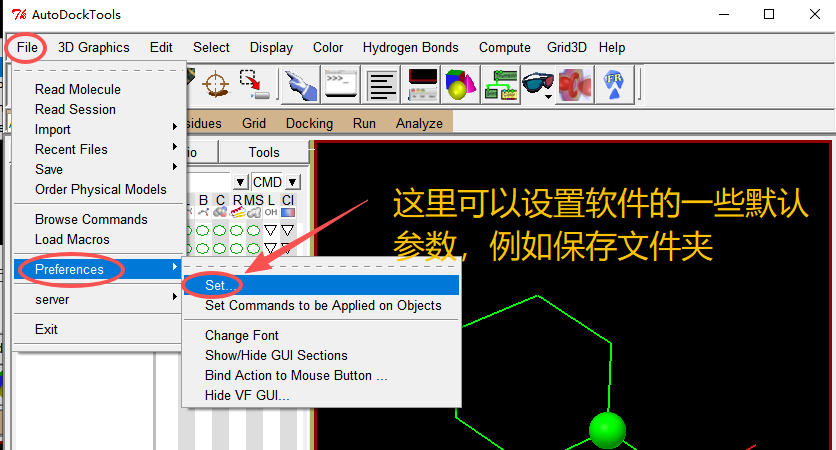
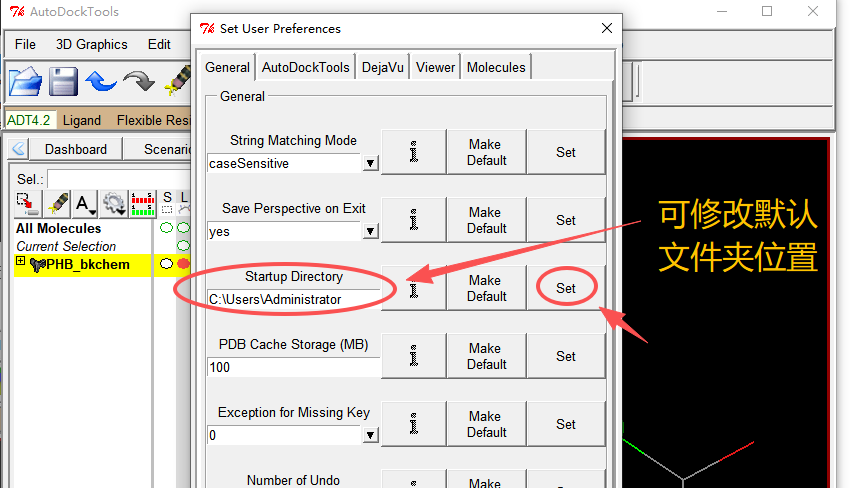
AutoDockTools(ADT)是完成蛋白、小分子预处理的核心工具,可以先设置一些基本参数,也可以不设置。
下面的默认的文件保存位置 “Startup Directory“ 就是软件默认的 PDBQT 文件保存的位置。
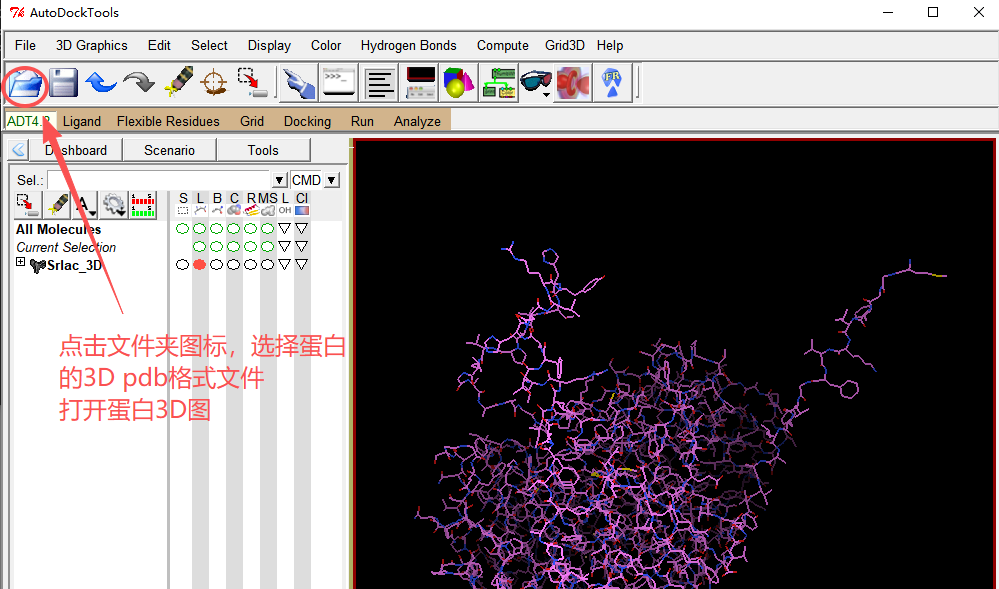
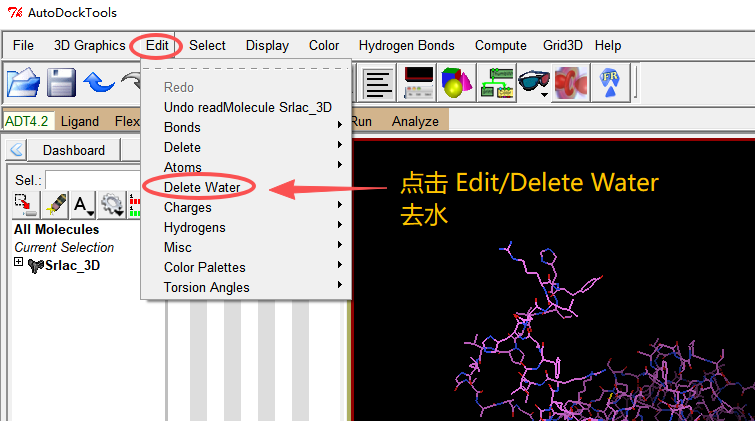
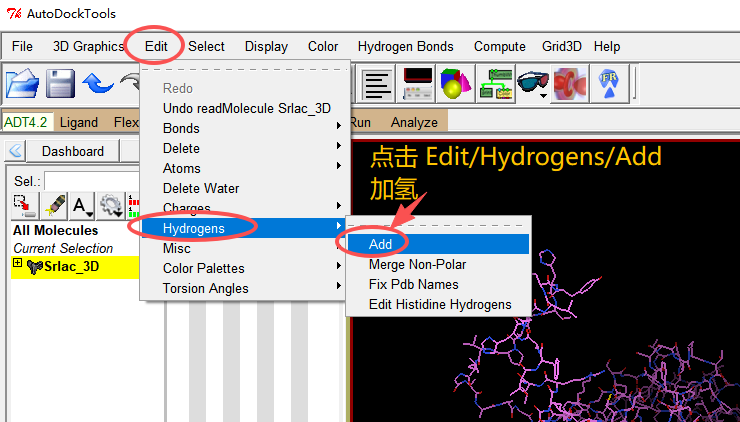
2、打开文件、去水、加氢。
点击 File/Read Molecule ,或者点击左上角的文件夹图标,在打开的窗口中选择蛋白的pdb文件,打开蛋白3D结构。
去水:点击 Edit/Delete Water,去掉蛋白的水分析。
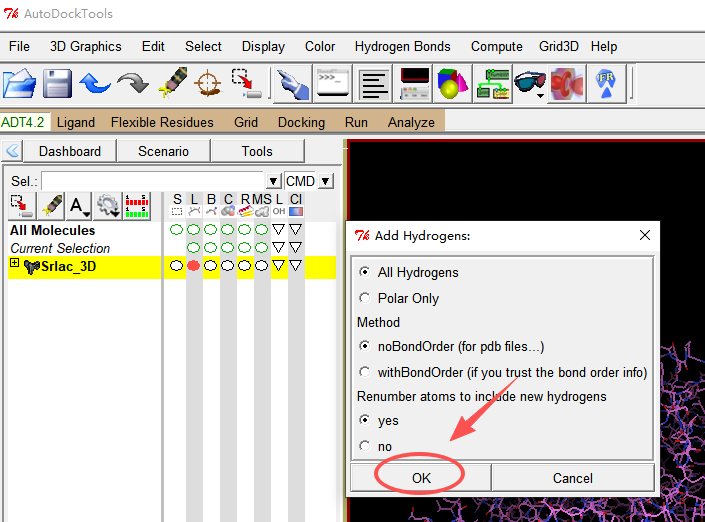
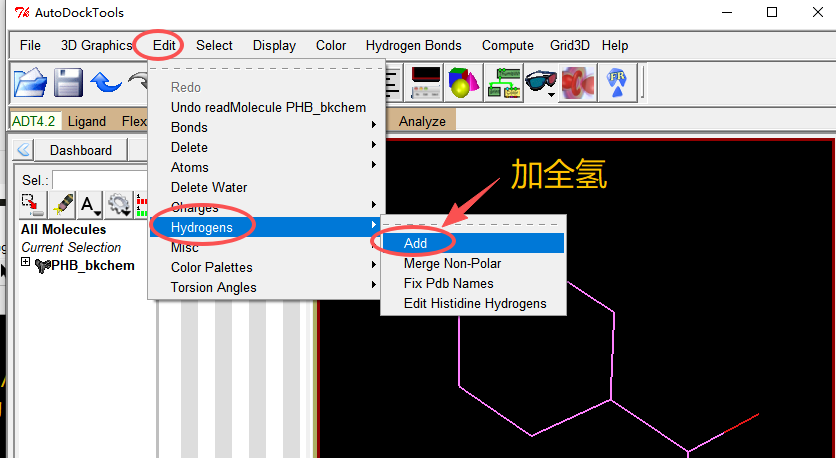
加氢:点击 Edit / Hydrogens / Add
蛋白的氨基(-NH3⁺)、羧基(-COO⁻)和羟基(-OH)需添加极性氢,以模拟生理环境下的电荷状态
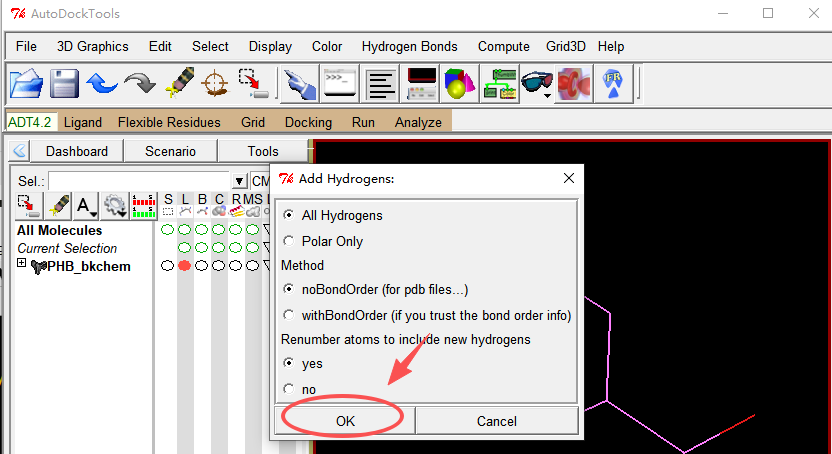
弹出对话框中不需要改默认参数(默认是加全氢),点击OK就行。
如果下载的蛋白中含有非功能配体分子(如晶体中的抑制剂)也需要提前删除,可以用PyMOL、ChimeraX等工具删除,另外非必须金属离子也需要删除(但是活性中心的金属离子需要保留)。
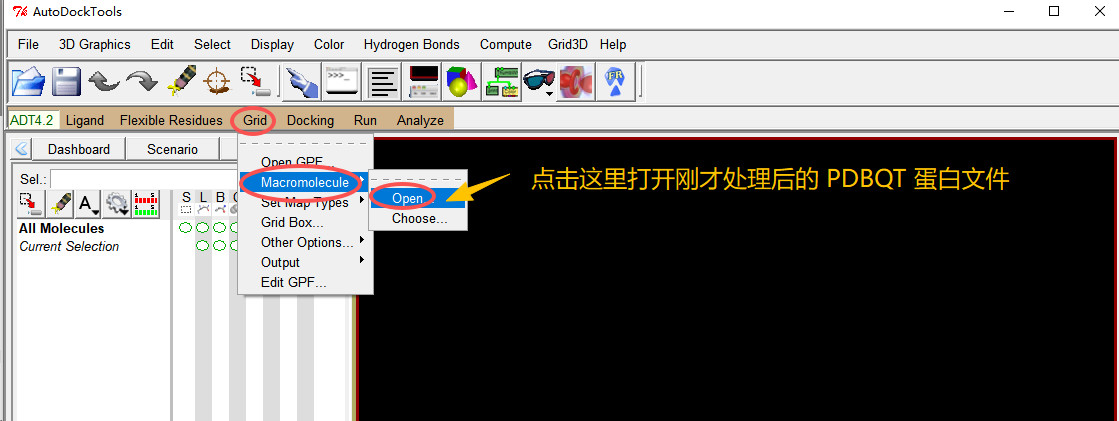
4、选择为受体文件,另存为 PDBQT 格式文件
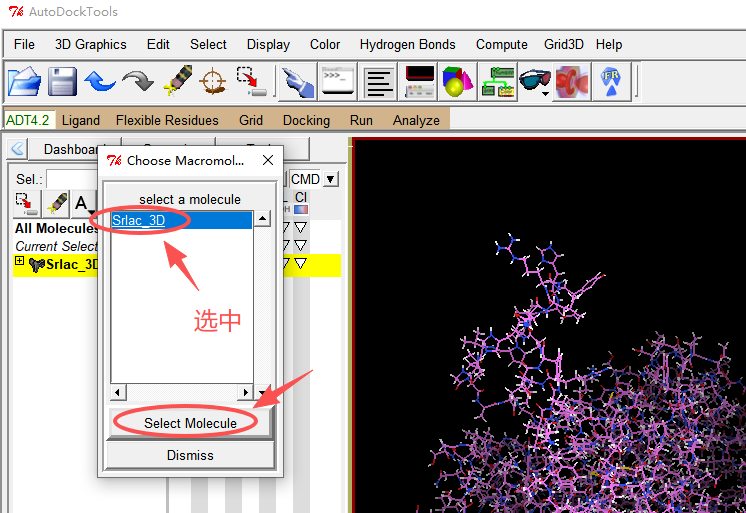
选择蛋白为受体:点击 Grid / Macromolecule / Choose,把蛋白保存为分子对接的受体。
弹出的对话框默认保存为 PDBQT,保存在软件默认的文件夹中。
1.3 配体(小分子)预处理
1、打开文件,加氢、设置为配体。
跟上面一样,打开配体分子的pdb文件。第一步跟蛋白一样,需要加氢。
把小分子选择为配体,这样后面才能操作。
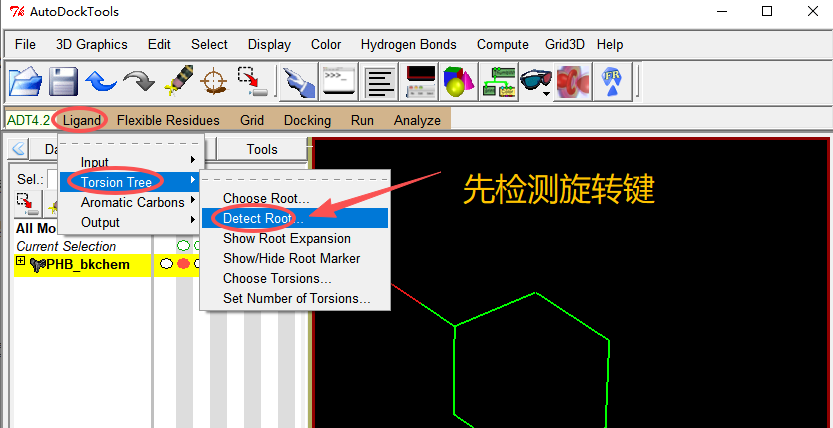
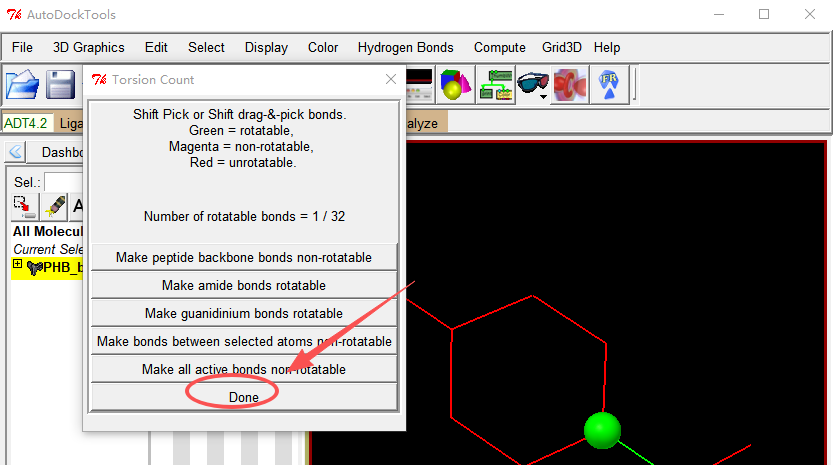
2、检测扭转键,选择扭转键
需要先检测旋转键,再选择旋转键。旋转键在分子对接的时候是可以旋转的,因为AutoDock Vina的分子对接是半柔性对接,小分子是可以转动的。
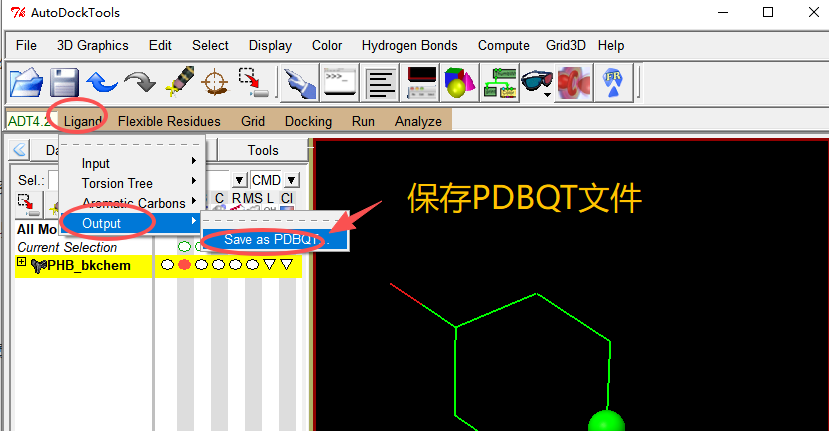
3、设置为配体,保存PDBQT格式文件
上面都设置完成之后,把小分子也保存成为PDBQT文件,也是保存到软件的默认的文件夹中。
1.4 对接Box和对接参数设置(重要)
确定对接的Box位置
分子对接首先要选择对接的盒子,也就是小分子与蛋白质对接的区域是什么。当然,如果无法通过文献或者其他的工具来获得蛋白的活性口袋位置的时候也可以把Box设置的非常大,让小分子与蛋白自由的进行对接。
- 如果有蛋白-蛋白配体复合物的晶体结构,那么以原配体所在位置作为活性口袋的中心;
- 如果没有原来的配体,通过文献能够知道活性口袋位置和关键残基,那么可以用PyMOL或者ChimeraX来测量残基的中心坐标;
- 如果文献也没有,可以直接利用AlphaFold3来预测小分子与蛋白的相互作用,将复合物中配体所在的位置作为Grid Box的中心(需要注意的是复合物通常要有较高的ipTM值才可信);
- 如果以上都不行,也可以通过在线工具来预测配体结合位点,例如 PrankWeb平台:https://prankweb.cz/,或者网站的本地版本 P2Rank:https://github.com/rdk/p2rank
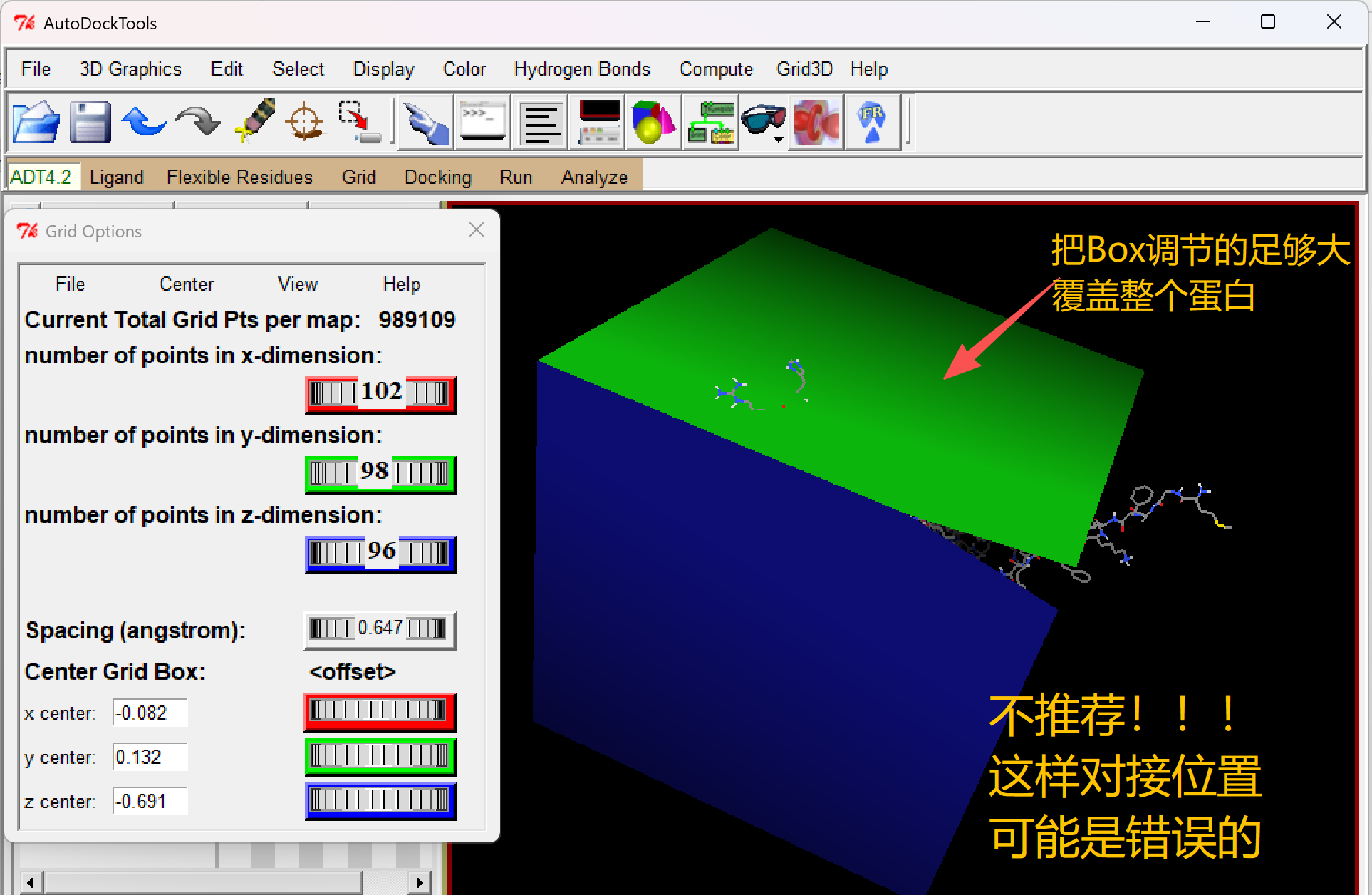
- 实在不行也可以先尝试用Box把蛋白全包裹,看看能对接上哪个位点再说。
- 寻找到了活性口袋,还需要用ADT来确认 Box 把所有的关键残基全部覆盖,避免配体被盒子边界截断。
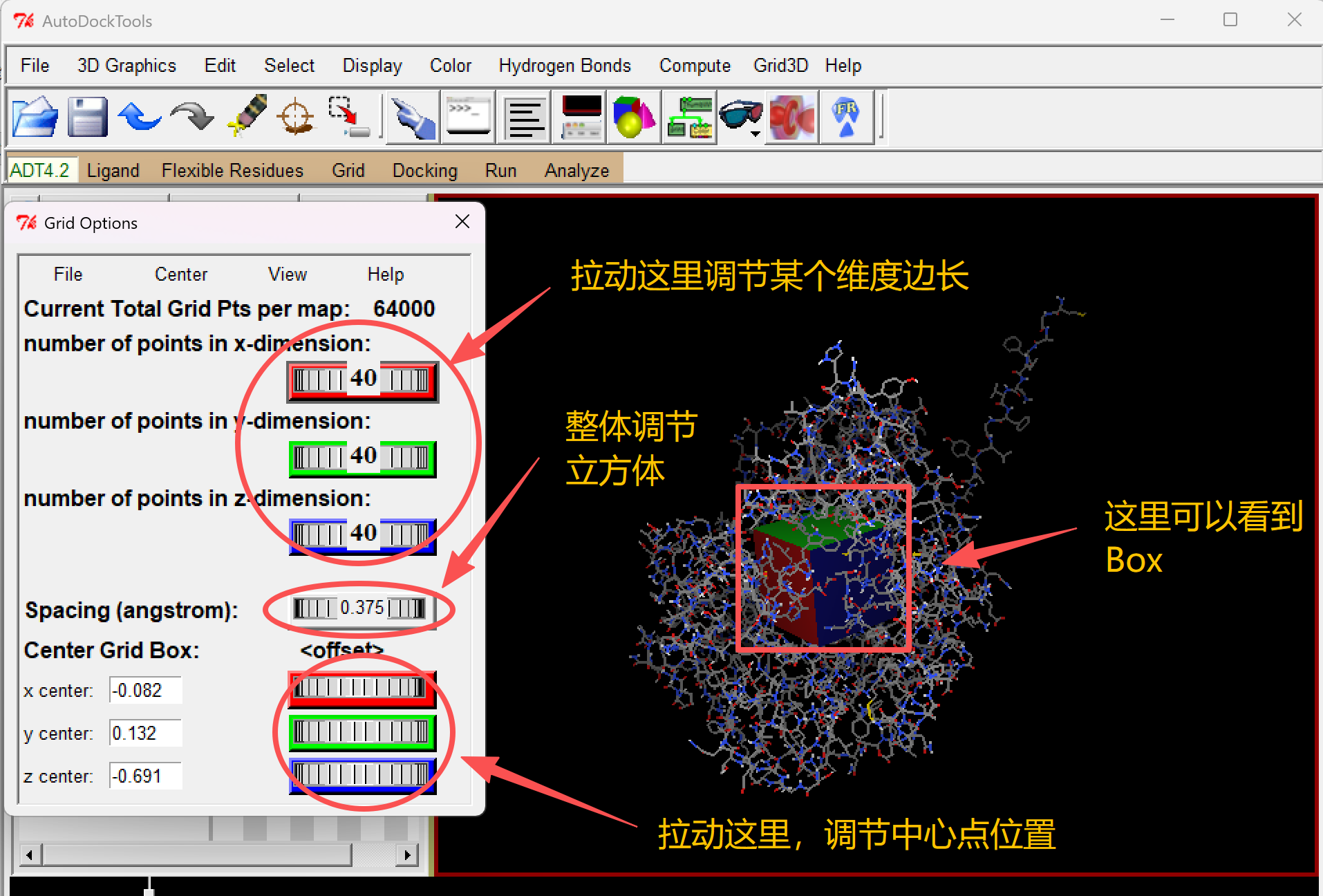
确定Box的大小
一般为 20×20×20 Å(建议直接设置为 30×30×30 Å),确保完全覆盖口袋,覆盖已知配体或关键氨基酸残基周围约5 Å范围。网格盒子过小会导致配体无法进入口袋,如果只对接几个分子的话,口袋大一些也可以,因为每次Docking也就是10s左右。
口袋如果覆盖区域太大会导致分子对接的位置发生错误,或者对接精度不够。如果太小了有可能配体无法自由的360度旋转,无法找到最优的姿态。
1.1、设置对接盒子:Box包裹整个蛋白
(不推荐这种方法,因为小分子会在多个口袋对接,不清楚哪个是正确的)font>
(1)打开处理后的蛋白的PDBQT文件
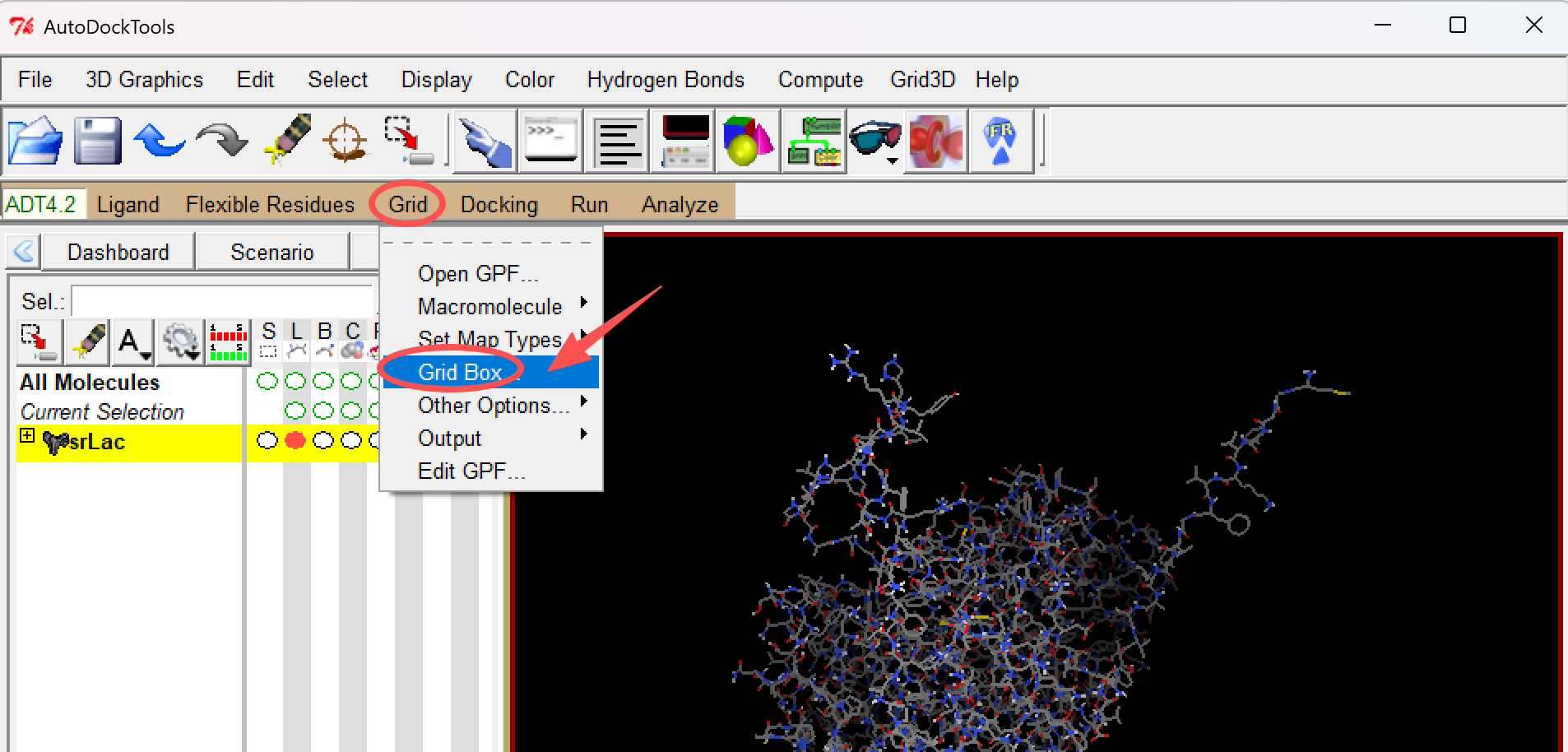
依次点击 “Grid / Grid Box” 打开用于对接的 Box。
在弹出的 “Grid Options” 窗口中,选择 Grid Box 的中心点和大小。
如果实在无法确定蛋白的活性口袋,那么可以把整个蛋白包裹。
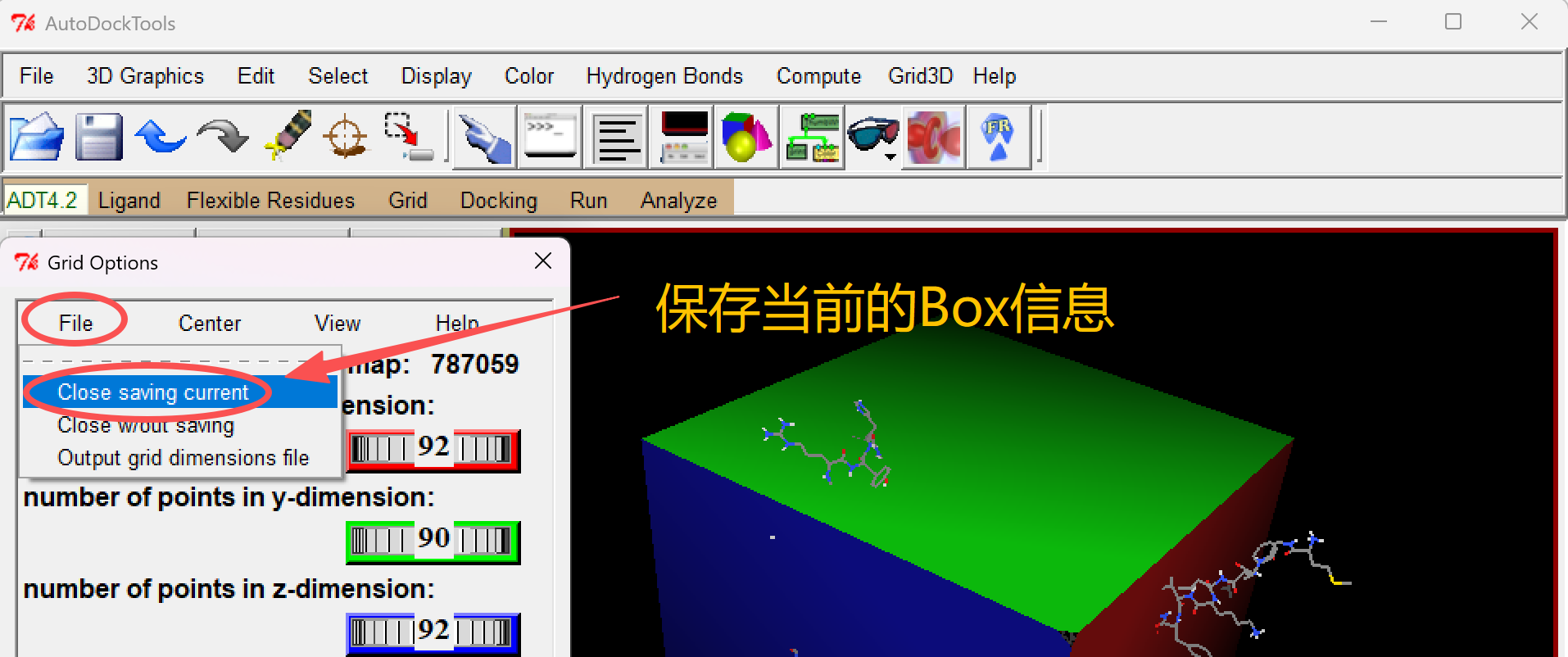
保存了 Grid Box 之后,可以利用 ADT 来保存用于Vina分析的contig文件。
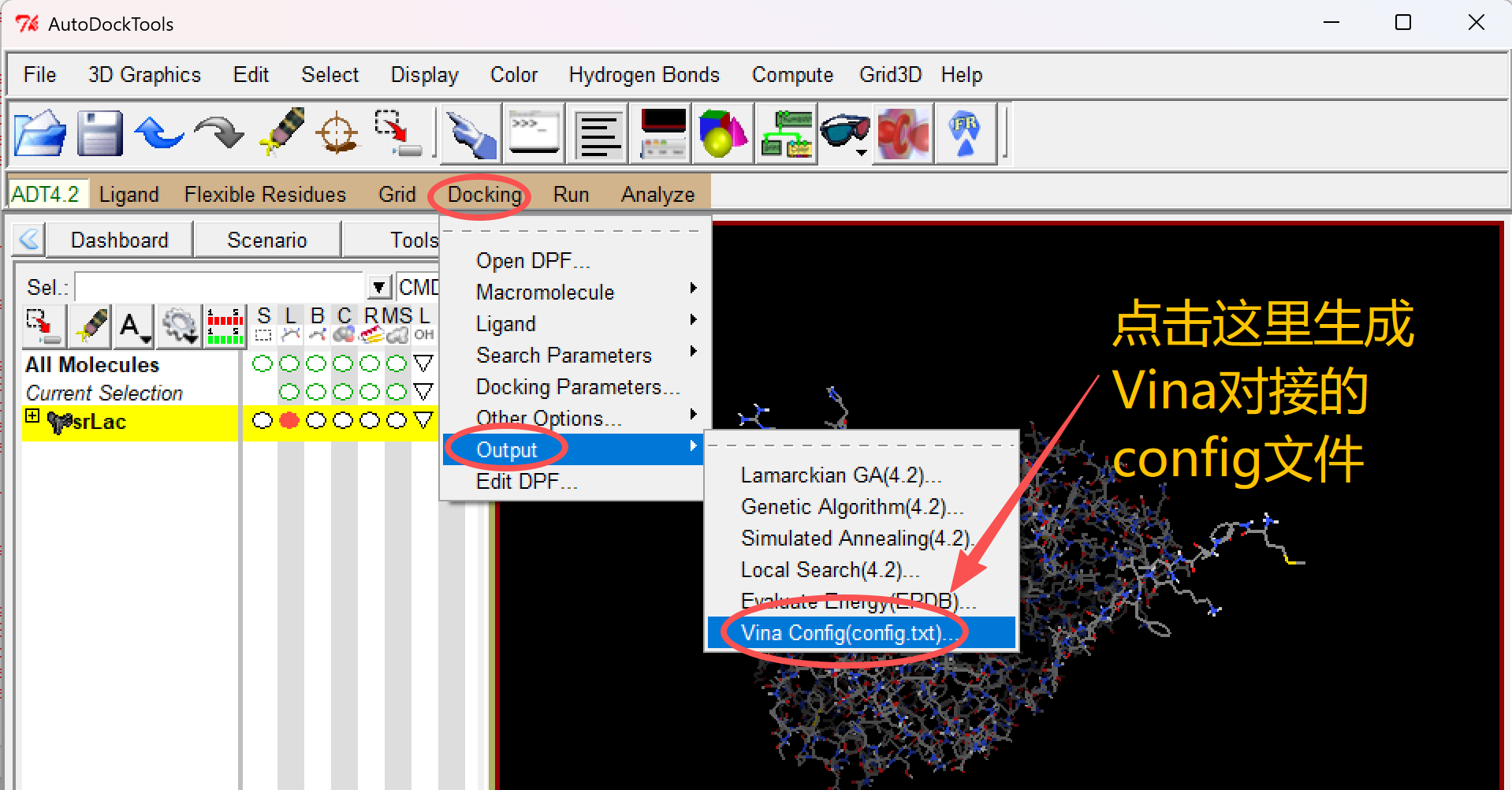
依次点击 “Docking / Output / Vina Config(config.txt)”
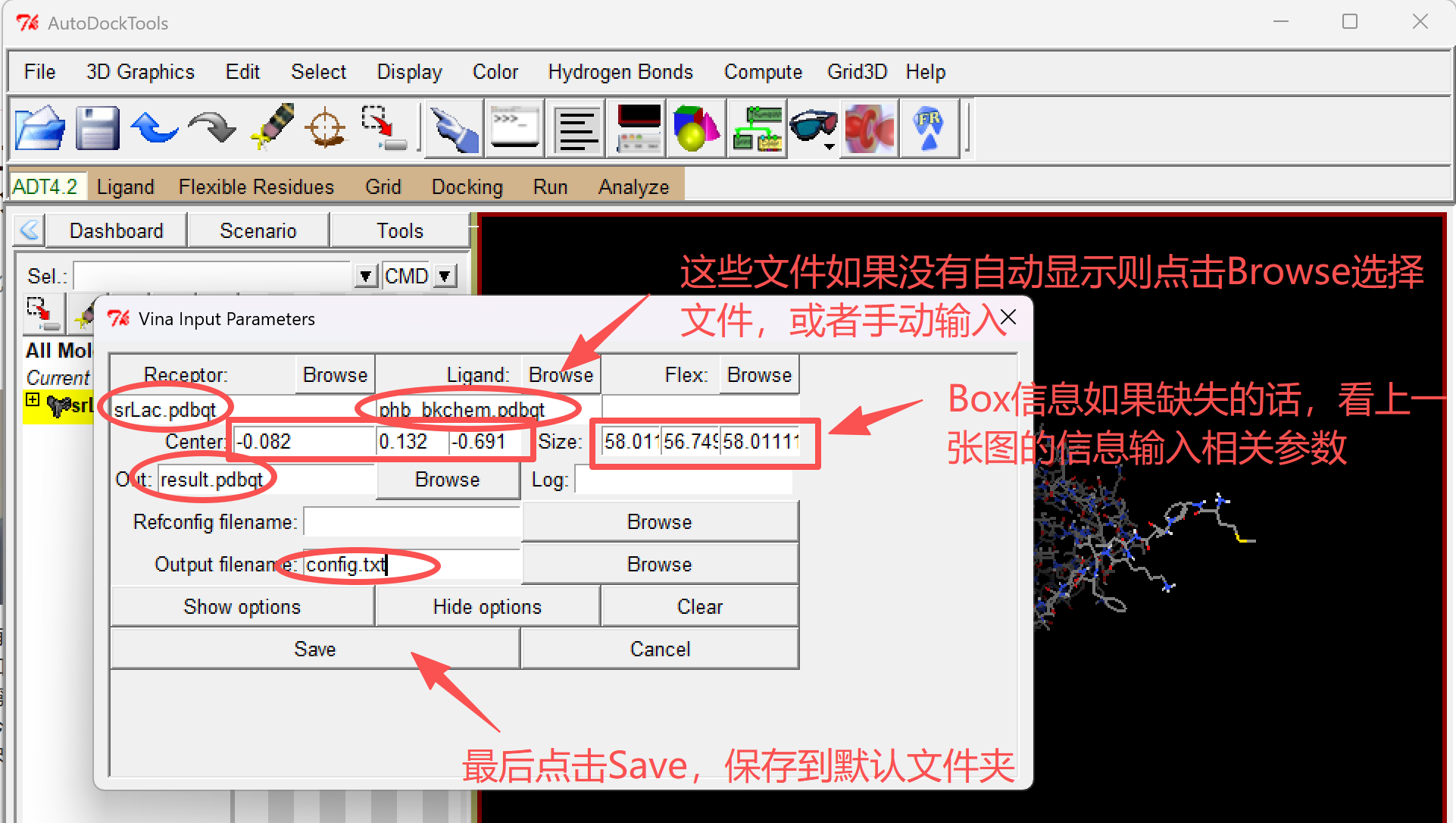
在弹出的 “Vina Input Parameters” 窗口中,需要输入 Receptor、Ligand、Out的文件名字,后缀都是pdbqt。
需要把对接盒子的 中心点和大小 给输入,Output filename默认是“config.txt”。
最后点击 “Save”,会默认的保存到ADT的配置的文件夹中。
1.2、设置对接盒子:通过 “蛋白-配体结构复合物” 来确定Grid Box
(1)从PDB下载带有配体的 蛋白-配体 复合物的晶体结构,或者通过 AlphaFold3 来进行蛋白-小分子分子对接得到“蛋白-配体复合物” 结构文件。
(2)利用ChimeraX打开 “蛋白-配体复合物”。
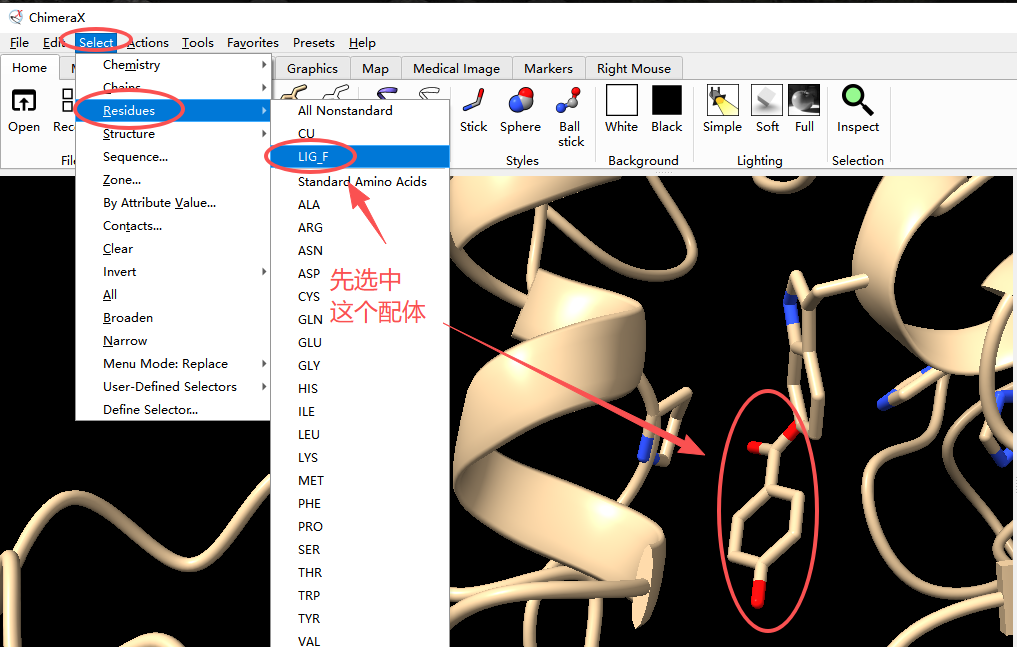
该复合物配体所在的位置就是活性口袋的中心,也就是我们后面要设置Grid Box的中心点,首先要选中这个配体。
点击 “Select / Residues / 配体名字”,这样就可以选中配体,选中后配体外面会有绿色的圈框起来。
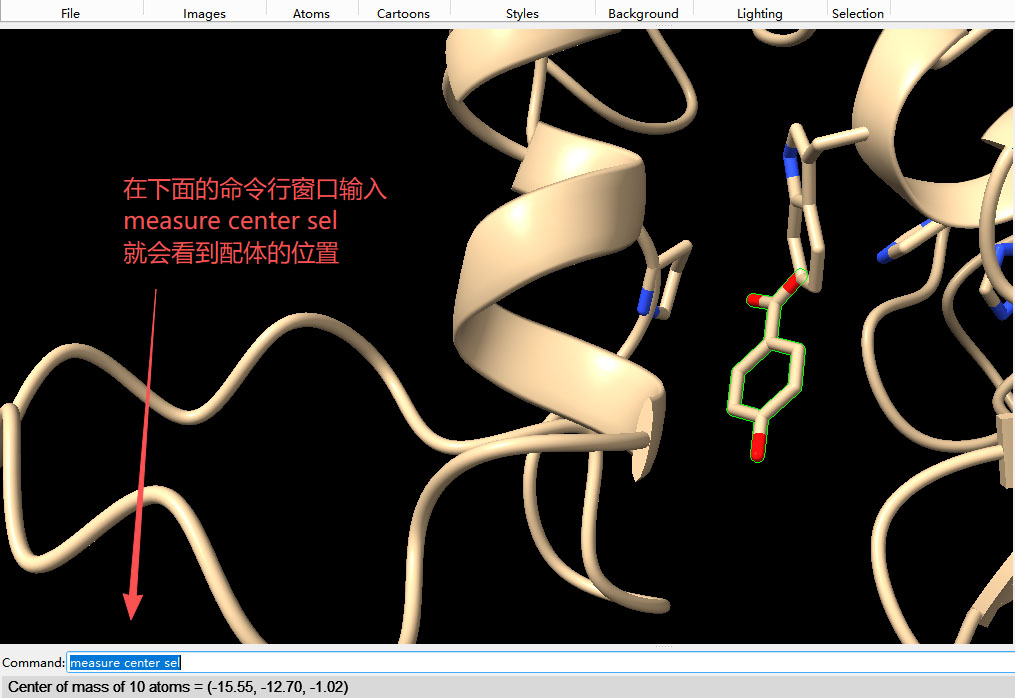
选中配体后,在下面的命令行中输入 “measure center sel”
点击回收后,下面就会出现配体的中心位置,这个就可以用于Vina 的Grid Box的中点。
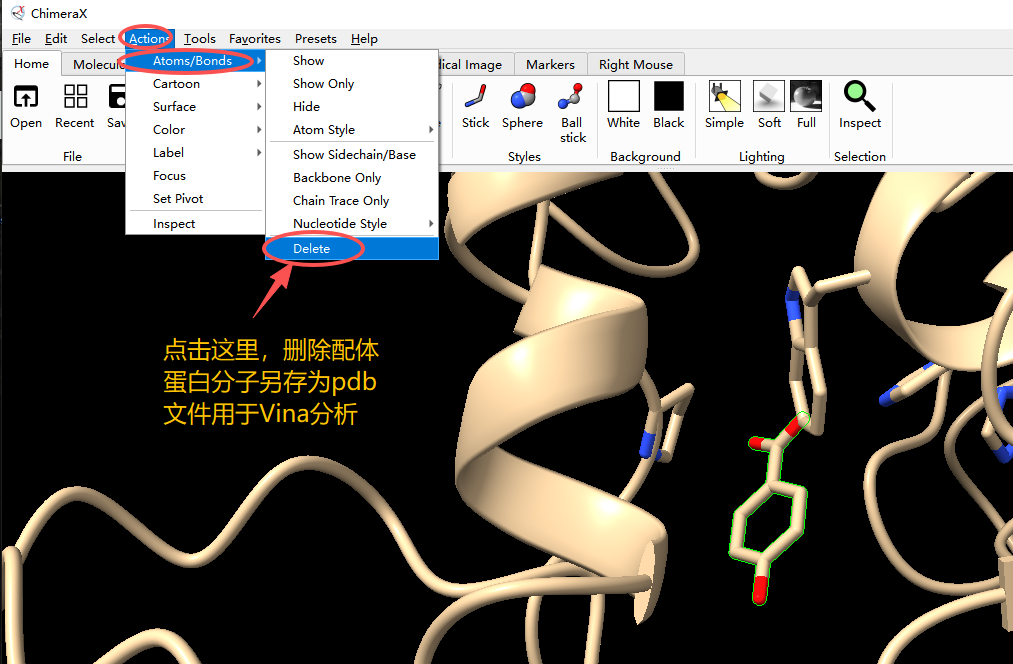
如果使用这个中心点,那么就要使用这个蛋白来作为Docking的受体蛋白分子。首先要把这个配体删除。
在选中这个配体的时候,点击 “Action / Atoms/Bonds / Delete” 就可以把配体删除了。再把这个蛋白另存为 PDB 文件格式就可以了,当然这个蛋白还是需要按照前面的步骤 “去水 - 加氢 - 设置为受体 - 另存为PDBQT格式”。
用这种方法来确定的中心点,最好还是按照上面 1.1 的方法,在蛋白上面测试一下是否的确包裹住了活性口袋,确定一个Box的边长是多少。
下面是ChimeraX和ADT展示的蛋白和Grid Box(变成为20Å),可以看到完全是一致的。
1.3、通过查找文献确定活性口袋关键氨基酸,对活性口袋中心定位,或者利用其他工具来预测活性口袋
根据上面介绍的在线网站自己尝试一下,今后有时间再更新。。。
2、修改配置文件
-
根据前面的方法,ADT可以生成一个 config.txt 文件,不过ADT生成的配置文件只有前面 9 行,还有一些其他的参数也可以在contig中进行设置,例如对接精度,输出的最优构想的数量,随机数种子等等。不添加这些信息也不影响分析,软件会使用默认参数或者随机数。
-
修改后保存为 “config.txt”,与受体、配体文件放在同一文件夹下,便于操作(因为毕竟下面的受体、配体都没有写出来绝对路径)。
receptor = receptor.pdbqt # 受体文件路径
ligand = ligand.pdbqt # 配体文件路径
center_x = 10.2 # 网格中心X坐标(在ADT中选择的)
center_y = 25.6 # 网格中心Y坐标
center_z = 18.9 # 网格中心Z坐标
size_x = 20 # 网格X方向尺寸(单位是Å,ADT中的Box大小 x 比例)
size_y = 20 # 网格Y方向尺寸
size_z = 20 # 网格Z方向尺寸
out = docking_result.pdbqt # 对接结果保存路径
exhaustiveness = 30 # 搜索 exhaustiveness(默认8,值越高越准确但耗时更长,如果只做几个分子对接,那么设置高一点)
num_modes = 10 # 输出最优构象数量(默认10,保留前3-5个即可)
energy_range = 3 # 最优构象与最差构象的能量差(kcal/mol),默认3
seed = 2077 # 随机种子,只要这个数值一样,重复分析做的结果都是一样的
Exhaustiveness:默认8,穷举度,数值越高,搜索越彻底,结果越稳定,但计算时间线性增加。对于高柔性配体,建议设为 32 或更高 。
Energy Range:1-4 kcal/mol(默认3)能量范围。规定了输出结果中,候选构象与最优构象之间允许的最大能量差。
1.5 分子对接
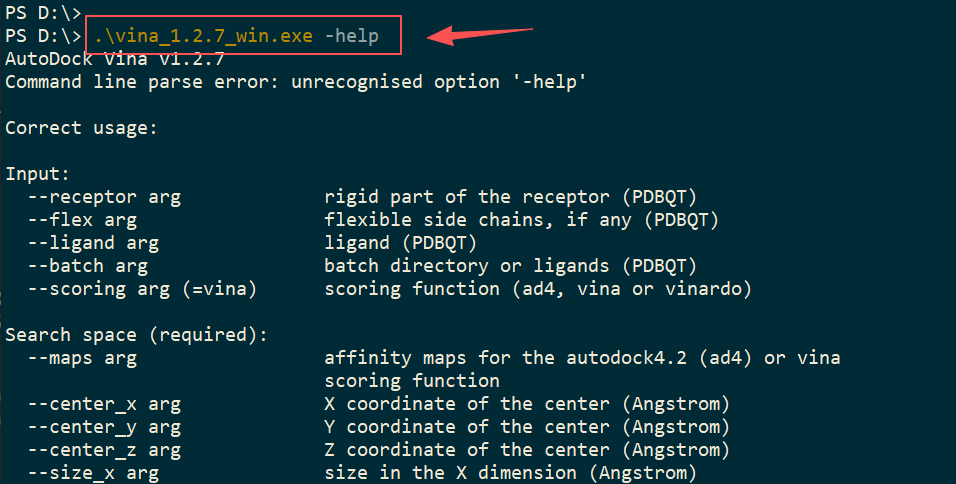
打开Windows的Powershell,进入“vina_1.2.7_win.exe” 和 pdbqt、contig.txt 所在的目录。
在命令行中输入:
vina --config config.txt --cpu 10 # 使用10个CPU线程,如果你的CPU只有8个线程,那就输入8
- AutoDock Vina基于OpenMP实现多线程计算,支持在多核系统上加速。多线程可以显著提高研究效率。现在电脑的速度都非常快,因此分子对接速度都很快,运行速度取决于小分子的大小,我们的对羟基苯甲酸分子数非常少,在高精度模式(exhaustiveness = 30)下也仅仅需要10 s就可以完成分析,如果是大分子则花费的时间可能会长一些。
运行的结果可以直接在对话框中看到,其中affinity这一列就是每组运行样本的亲和能,也就是最终评估是否有相互作用的最核心的参数。
Performing docking (random seed: 2077) ...
0% 10 20 30 40 50 60 70 80 90 100%
|----|----|----|----|----|----|----|----|----|----|
***************************************************
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.724 0 0
2 -5.684 0.104 1.498
3 -5.67 2.015 4.318
4 -5.316 20.69 22.72
5 -5.258 24.82 25.21
6 -5.25 24.28 25.17
7 -5.097 23.54 25.21
8 -5.086 21.06 22.84
9 -5.017 25.87 26.42
10 -4.92 0.9608 4.38
运行完成后就会在文件夹中看到一个结果文件(文件名跟contig.txt中的一致),这就说明分析成功了,可以用于后续的分析和可视化。
1.6 结果分析和可视化
1、Affinity(亲和力得分)
最关键的指标,单位是kcal/mol
| Affinity分值 | 解读 |
|---|---|
| < -9.0 kcal/mol | 表示配体与受体有极强结合 |
| < -7.0 kcal/mol | 通常认为具有较好的结合潜力 |
| -5.0 ~ 0 kcal/mol | 结合较弱,可能存在空间碰撞或理化性质不匹配 |
| 正值 | 表示小分子与蛋白存在严重的范德华排斥,结合极不稳定 |
2、RMSD(均方根偏差) ,用于衡量构象间的结构差异
- RMSD < 1.0 Å:几乎相同构象
- RMSD < 2.0 Å:通常认为属于同一结合模式(“cluster”)
- RMSD > 3.0 Å:明显不同的结合姿态
如果采用蛋白全包裹的Grid Box,RMSD相差太大就说明了对接了不同的活性口袋。
3、PDBQT 文件(构象文件)
该文件包含了 Vina 预测出的多种候选对接姿态(Binding Poses)。PDBQT 在 PDB 格式基础上增加了部分电荷(Q)和 AutoDock 原子类型(T)信息 。
通过PyMOL或ChimeraX可以可视化对接结果,评估构象合理性,分析配体与受体之间的相互作用(可视化参考其他教程)。
二、AutoDock Vina分子对接软件安装和准备
2.1 分子对接流程和所需软件列表
下面是一些关键的软件列表,其中MGLTools(含ADT)和AutoDock Vina是核心软件,其他的都是辅助软件,很多步骤都可以由多个软件实现相同的功能,这里只列出来免费开源的软件。大家也可以使用自己熟悉的软件。
| 软件名称 | 核心功能 | 备注 |
|---|---|---|
| MGLTools 开源免费 |
1. 蛋白去水、去除冗余配体 / 杂质; 2. 添加氢原子、计算 Gasteiger 电荷; 3. 生成蛋白 pdbqt 文件; 4. 定义对接网格(结合口袋) |
集成了AutoDockTools (ADT),安装程序自带 Python 2.7解决python环境依赖问题 |
| AutoDock Vina 1.2.* 开源免费 |
核心分子对接工具,执行蛋白 - 小分子分子对接计算,输出结合能及构象排名。支持 GPU 加速、更大分子对接,兼容更多格式。 | |
| OpenBabel 开源免费 |
1. 分子格式转换(mol2→pdb→pdbqt); 2. 配体构象优化(MMFF94 力场); 3. 计算 Gasteiger 电荷 |
核心格式转换工具,可替代 ADT 完成配体 pdbqt 文件生成,支持批量处理 |
| Avogadro 开源免费 |
可视化软件,主要用于构建、优化、分析三维分子结构,可以将小分子的2D结构进行立场优化后转化成为3D结构。 | 格式转化工具,可视化操作界面,操作简便 |
| ChimeraX 开源免费 |
1. 多构象叠加分析; 2. 结合口袋氢键网络绘制; 3. 配体与蛋白相互作用量化(如距离测量) |
PyMOL的开源替代工具 |
2.2 MGLTools安装(含ADT)
1、进入MGLTools官网:https://ccsb.scripps.edu/mgltools/
2、点击左侧的Downloads:https://ccsb.scripps.edu/mgltools/downloads/
3、点击下载最新版本的工具,我们这使用的是Windows系统,所以点击下载Windows系统的安装软件。
现在写这个教程的时候最新版本是1.5.7。
4、安装过程就是一路点击 “下一步” “Accept” 就行了。
注意:
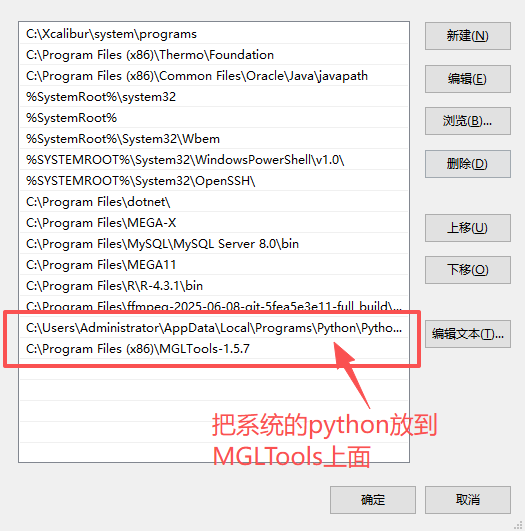
需要注意的是MGLTools并不是不依赖python 2.7,而是在安装包中整合进了python 2.7的运行的依赖。并且在安装过程中会把MGLTools的工具放到环境变量里面。如果系统里面安装了其他版本的python,可能python会被替换成为2.7版本。因此需要重新添加系统的python路径到环境变量里面,而且要放到MGLTools上面。
*2.3 AutoDock Vina 1.2.下载
1、进入AutoDock Vina的官网
AutoDock Vina:https://vina.scripps.edu/
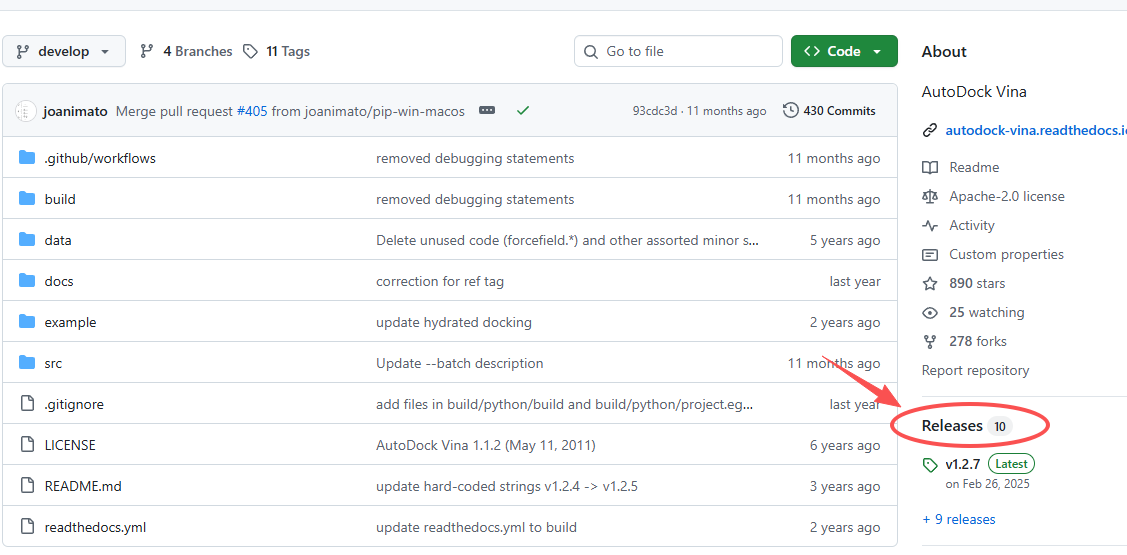
GitHub主页(从这里下载):https://github.com/ccsb-scripps/AutoDock-Vina
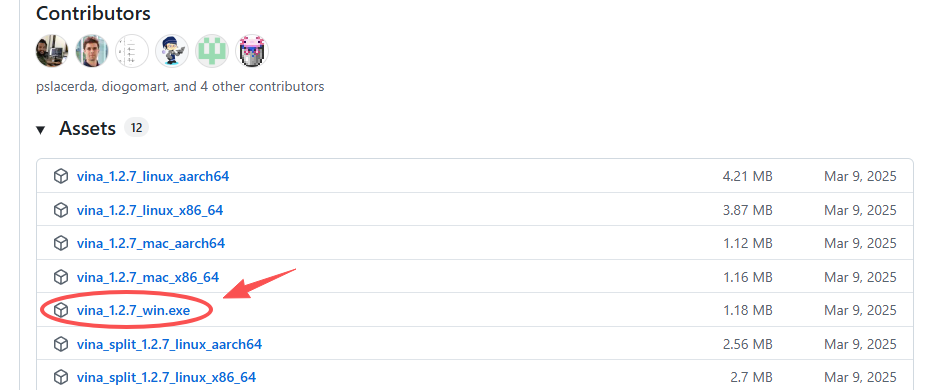
当前最新版本是2025年5月发布的 V1.2.7 版本,点击下载就行。
下载后是一个exe文件,由于AutoDock Vina是一个命令行的工具,并没有图形界面软件,所以不要尝试双击安装。将这个exe文件放到一个目录下面,就可以用powershell等工具访问运行了。
2.4 Open Babel安装
1、进入GitHub主页下载
进入Open Babel官网:http://openbabel.org/
进入Open Babel的GitHub官网(从这里下载,点击右侧的Releases):https://github.com/openbabel/openbabel
2、安装软件
安装过程就是一路点击 “下一步” “Accept” 就行了。
2.5 ChimeraX
ChimeraX的安装参考另一个教程:
3D分子可视化:ChimeraX的安装与使用
2.6 其他工具软件
1、免费开源的小分子结构绘制软件BKChem
从官网下载:🔗https://bkchem.zirael.org/index.html
点击左侧的Download进入下载页面,下载Windows的 “.exe“ 版本,直接双击安装就行。
2、免费开源的分子可视化软件 Avogadro
官网:🔗https://avogadro.cc
进入官网,点击顶部的 Downloads,下载后点击下一步安装就行。








评论区