原创内容,欢迎转载,转载请注明出处
主笔:于浩
导读
1、截止到2025年底的时候,开源的能够预测蛋白与蛋白、蛋白与DNA相互作用的最好的软件就是 AlphaFold3 和 RoseTTAFold All-Atom 这两个软件,这两个软件都是开源的,本教程简单的介绍一下 AlphaFold3 预测蛋白与蛋白/DNA的相互作用。
2、AlphaFold3相对于AlphaFold2提升的部分实际上主要是对其他分子结构的预测以及分子间相互作用的预测能力的增加。是一次从“蛋白质结构预测”到“全生物分子相互作用建模”跃迁,被誉为“理解生命分子语言的新工具”。
因此除了基本的蛋白质结构预测之外,本网站会逐步的把利用AlphaFold3来进行分子相互作用的使用添加到网站上。
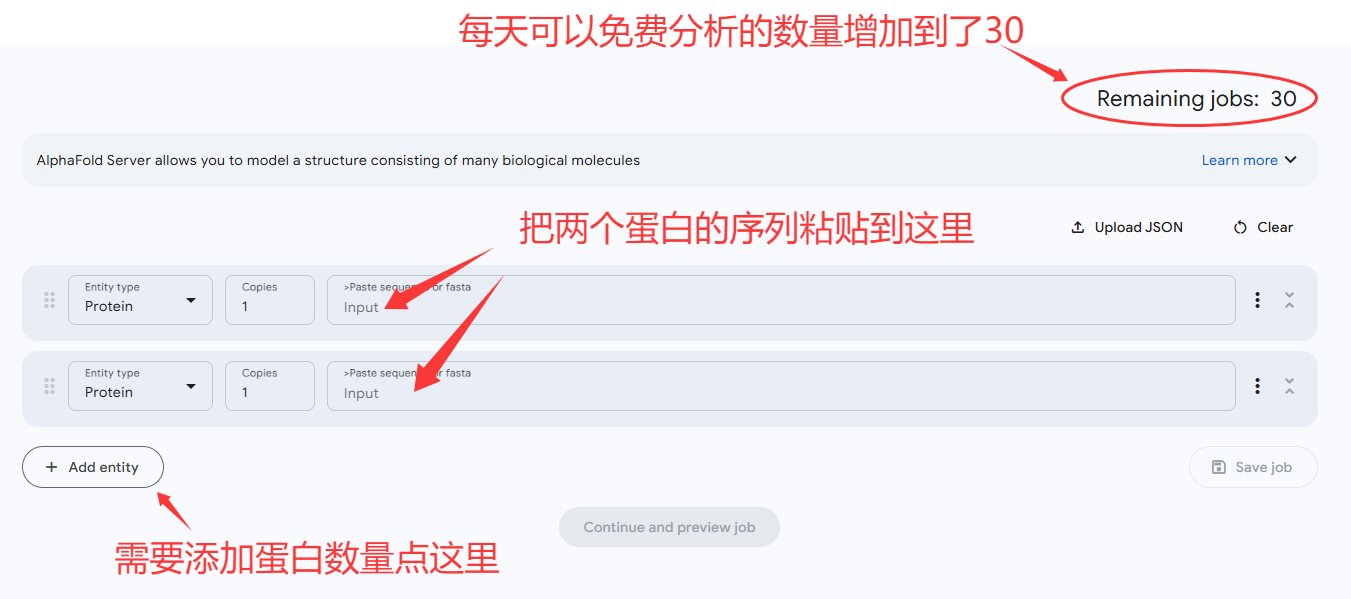
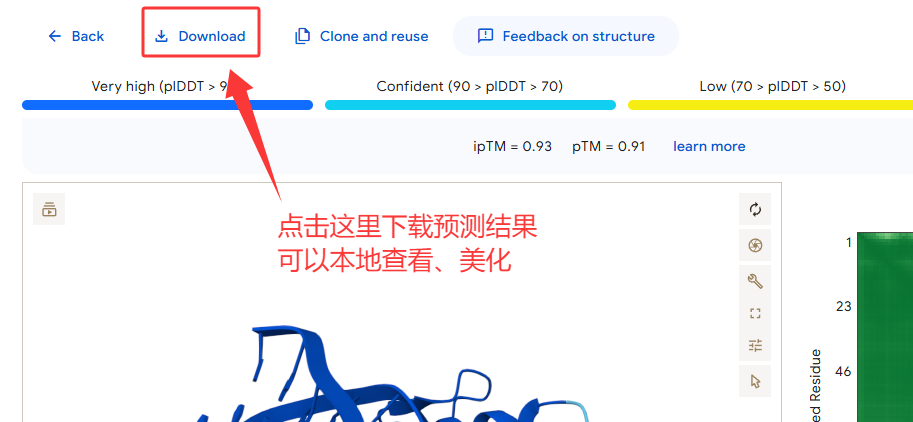
3、使用Alphafold Server在线预测每天可以预测30个,但是需要先解决登录网站的问题,如果计算量比较少的话,建议使用在线服务器。本地运行要达到在线服务器的速度需要的GPU计算资源太多了。一般来说在线运行的数量足够我们用了,如果一个账号不够用可以多申请几个账号,很多专门的做蛋白结构的科研团队也是这么做的。
本教程的例子在在线服务器只需要6 min,但是我们自己的小服务器上面跑用了46 min。
一、利用AlphaFold3进行“蛋白-蛋白”相互作用预测
(一)、AlphaFold3在线服务器进行 蛋白-蛋白 互作研究
1、登录
首先要打开AlphaFold3服务器,需要科学上网和Google账号。
2、蛋白序列
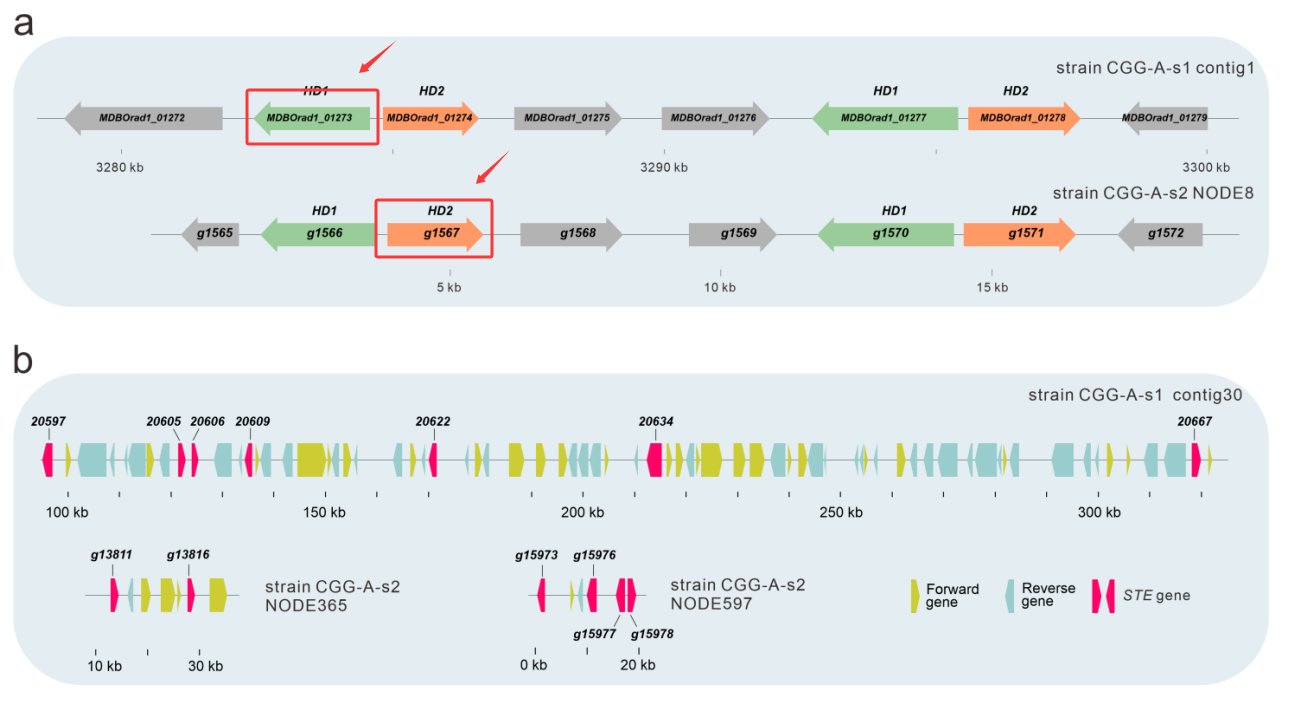
这里使用的蛋白序列是我们从团队之前发表的一个文章(J Fungi, 2023, 9:266; https://doi.org/10.3390/jof9020266)中选择了2个序列。团队首次报道了长根菇的全基因组序列,并且测定了两个交配型不同的孢子单核体,下图是Mating A和Mating B位点的结构。根据文献报道可亲和的Mating A位点中,位于同源染色体上面的HD1和HD2会形成异二聚体。
因此本教程选择了下图中红色框中标记的两个蛋白,利用AlphaFold3预测蛋白结构。序列也可以从实验室网站上面下载:
https://file.mushroomlab.cn/genome
>MDBOrad1_01273
MSTIHDRLVSAIDSLLHISGGPSLDEFAAAYTDSIDPSTELLEDENALPYDLAADIDFVCQSLMEASLADDEWLGRLTQDLGVVKTHLIPFQEESISSPSRNSSPTPSSSPSRESSPTPSRRASPQPSRATSPTAVDQQPLADSKDVPPTQESEVPTIPVPPTLSSRVRVPAPLTTTESDPTSVAGPSSRSPSPSPSTDTQPPTPTTPTSSPTFIQLLLDHLVANPYNPYPHKSFLSHVATSTSTSEKDVTTYLRSLRSKLGWNRIQKRHFPKPQWTQWQMIAWLEGLWRFEHSQQPNFEEWDKYQGPDGLGGKMNGLDAELAVVVHELERMAERREGPTEVAKVLDQVNWEEFVEKKNLKRKAEEDEASHPRVVSEPTKKRPRSESEESQVVSEPSKKRSRSESPESDDGTPPPPSKRHCSLPTMSLSTPPELTCWQTSAWVPPAQVSLNTFSYDRYTVPVLVDDESEDGSEGGSVGREWLLLCFWFLFSVALFGFPILRIAGFGLLASTPETADHISLPSWDFSSYVPQSQPATLSVSTLPDVSFPELLNLDLFSPAYTSTGKDLPSATPAMDHYIPIQPLTDDLPFDFDSQSWSFNYDGLTLDMGVDWSSLGVEGV
>g1567
MTANTRSDLLNRALRIAQRFKETVGPSCVNPTSRKPAANITIPTLNLELPADVQAELADTTAHQELRDTLRREITGSQAEYQRIYDTACQRALTSPSPQLHALGALSKLLTRRFQTQWIPHVLKQYLSTKEEIARSIQAASTIPSTSSKAFNHAFIPILTAYFMWNAFPPAADKRVLASKGLMTDRQIDVWFQNRRALARKHGVRLEKAAYKAKTRNGVPAELMKQITEFTSFSSPNHQQVRKDDEERQWRWNVGWGSLGLSGLWERGRIVAHSILEKNKQDIKVKAVDVLAMHSPLEDAPQSMEEWASLRKEGKVVFDYAWLKAPYAQRTTVVPSPAPPVPDPHPSEQGEVSLRRVTEMFADAGRIHFGLLGVRNEGEAGSDYGVGAATMGVTRDVRWYVSRIGSWNAPRSRPEVREVEVKAKKRKSPEKKEKKRLGERRQPTSRYEPYPGSSRSSSKSSLRSQGSSSSLSSSSSRTPSLTSASSDEEDDMESLFGGGVQLDLEGFTPVGNGKGMMNPSAGMMTDPYIPMGMDIHGMGYGYSNLDLGMNYGSWGLLGDVF
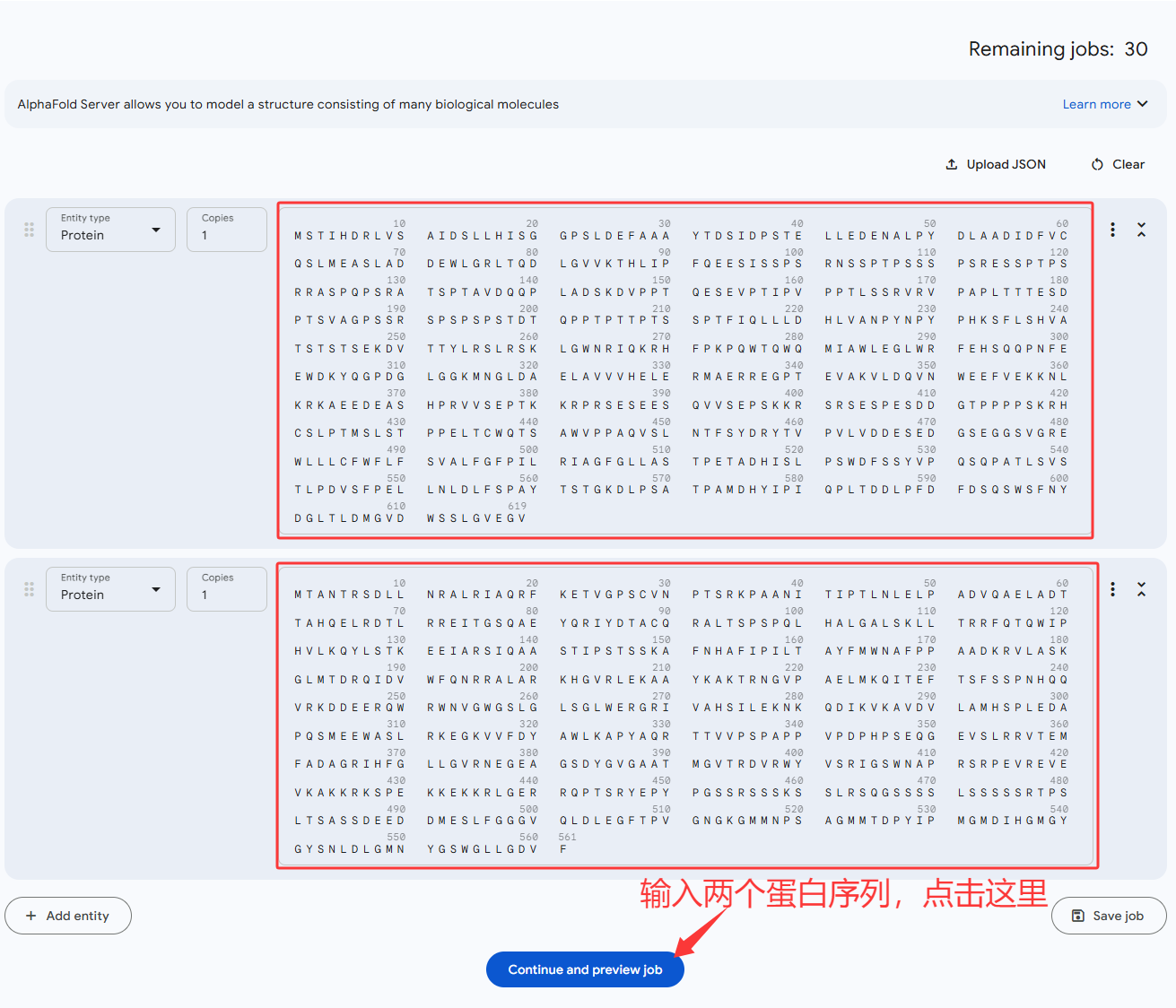
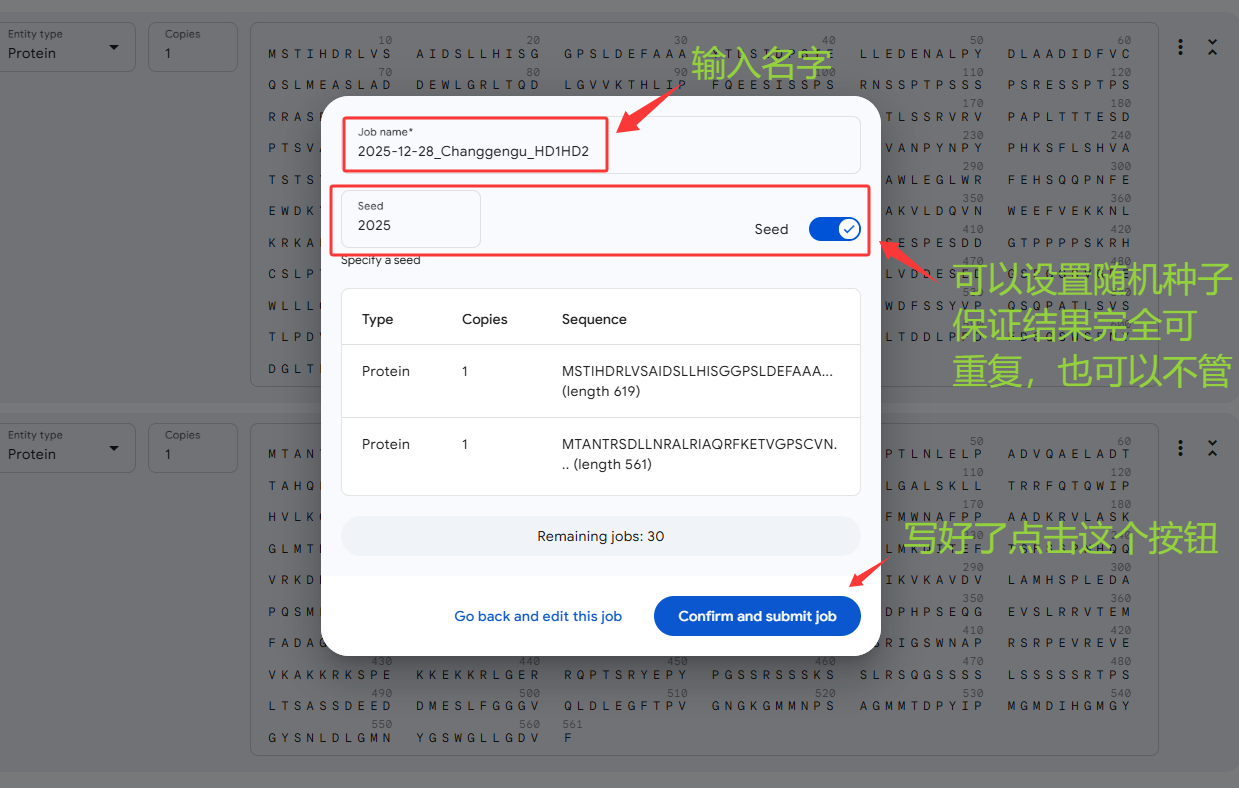
2、在下面的输入框里面输入两个蛋白的序列,之后点击下面的 “Continue and preview job”。
这个时间也不是特别长,10 min内就能出结果,所以大家能使用在线服务器就使用在线服务器。
(二)、蛋白-蛋白 互作结果分析
查看结果
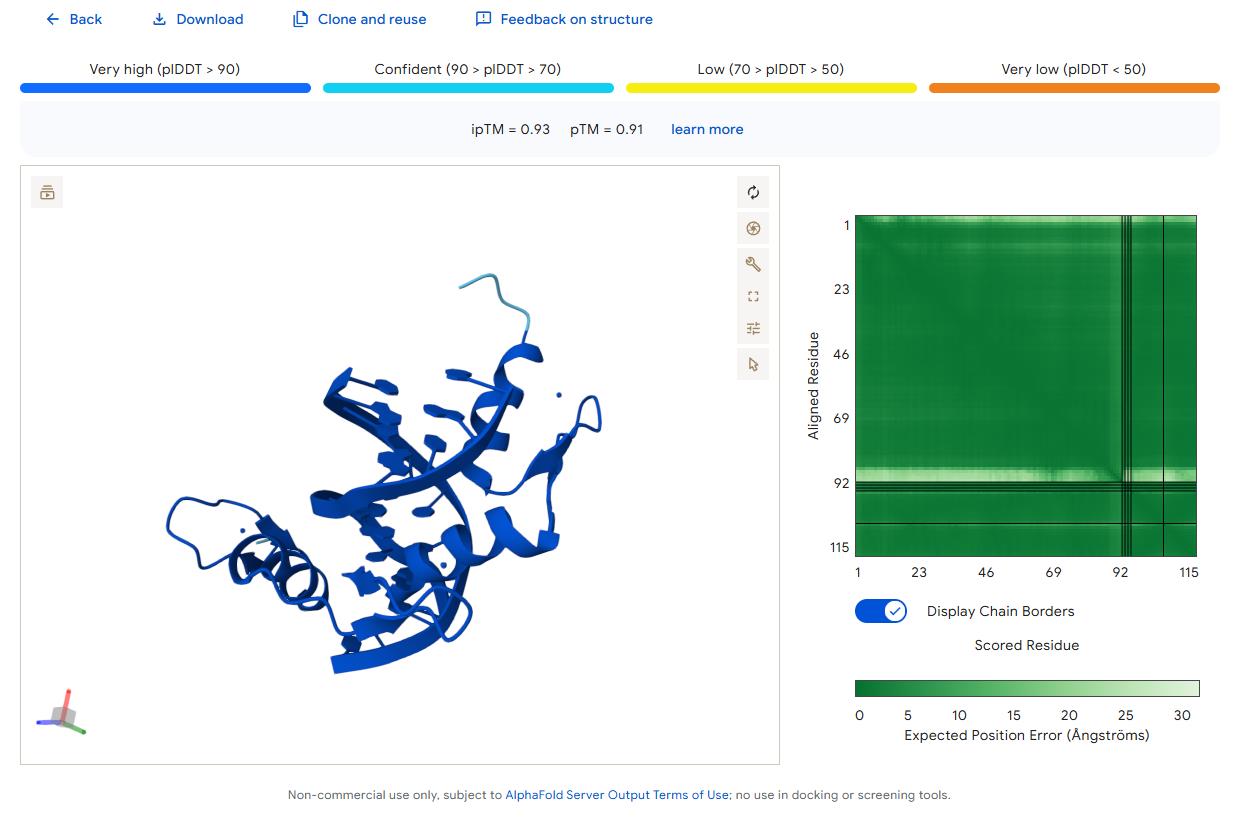
下图就是AlphaFold3服务器给出来的最好的结果,但是从给定的参数看,实际上的效果并不好。
实际上这个也比较好理解,我在分析之前在NCBI上面利用Blastp在PDB数据库中比对了一下相似序列,结果显示MDBOrad1_01273在PDB数据库中无相似性的序列。g1567在PDB数据库中比对分数最高的Query Cover只有8%,相似性只有36.17%。因此可能是因为AlphaFold3没有该类蛋白的训练数据导致蛋白结构预测的结果就不准确,后面的分子对接不准确就很容易理解了。
(三)、在本地Linux服务器进行“蛋白-蛋白互作” 分析
AlphaFold3工具本地部署可以参考本网站另外一个教程:AlphaFold3:利用AlphaFold3进行蛋白质三级结构预测&AlphaFold3的本地安装。
AlphaFold3的 GitHub官网 也介绍了Docker容器方法和Singularity方法进行安装使用,也可以直接根据官网的指示进行安装。
1、文件准备
json格式的蛋白输入文件
根据官网给出来的标准格式进行修改的,下面只是最简单的格式,详细的格式看官网的教程:AlphaFold 3 input格式
{ "name": "propro_MDBOrad1_01273_g1567", "sequences": [ { "protein": { "id": ["A"], "sequence": "MSTIHDRLVSAIDSLLHISGGPSLDEFAAAYTDSIDPSTELLEDENALPYDLAADIDFVCQSLMEASLADDEWLGRLTQDLGVVKTHLIPFQEESISSPSRNSSPTPSSSPSRESSPTPSRRASPQPSRATSPTAVDQQPLADSKDVPPTQESEVPTIPVPPTLSSRVRVPAPLTTTESDPTSVAGPSSRSPSPSPSTDTQPPTPTTPTSSPTFIQLLLDHLVANPYNPYPHKSFLSHVATSTSTSEKDVTTYLRSLRSKLGWNRIQKRHFPKPQWTQWQMIAWLEGLWRFEHSQQPNFEEWDKYQGPDGLGGKMNGLDAELAVVVHELERMAERREGPTEVAKVLDQVNWEEFVEKKNLKRKAEEDEASHPRVVSEPTKKRPRSESEESQVVSEPSKKRSRSESPESDDGTPPPPSKRHCSLPTMSLSTPPELTCWQTSAWVPPAQVSLNTFSYDRYTVPVLVDDESEDGSEGGSVGREWLLLCFWFLFSVALFGFPILRIAGFGLLASTPETADHISLPSWDFSSYVPQSQPATLSVSTLPDVSFPELLNLDLFSPAYTSTGKDLPSATPAMDHYIPIQPLTDDLPFDFDSQSWSFNYDGLTLDMGVDWSSLGVEGV" } }, { "protein": { "id": ["B"], "sequence": "MTANTRSDLLNRALRIAQRFKETVGPSCVNPTSRKPAANITIPTLNLELPADVQAELADTTAHQELRDTLRREITGSQAEYQRIYDTACQRALTSPSPQLHALGALSKLLTRRFQTQWIPHVLKQYLSTKEEIARSIQAASTIPSTSSKAFNHAFIPILTAYFMWNAFPPAADKRVLASKGLMTDRQIDVWFQNRRALARKHGVRLEKAAYKAKTRNGVPAELMKQITEFTSFSSPNHQQVRKDDEERQWRWNVGWGSLGLSGLWERGRIVAHSILEKNKQDIKVKAVDVLAMHSPLEDAPQSMEEWASLRKEGKVVFDYAWLKAPYAQRTTVVPSPAPPVPDPHPSEQGEVSLRRVTEMFADAGRIHFGLLGVRNEGEAGSDYGVGAATMGVTRDVRWYVSRIGSWNAPRSRPEVREVEVKAKKRKSPEKKEKKRLGERRQPTSRYEPYPGSSRSSSKSSLRSQGSSSSLSSSSSRTPSLTSASSDEEDDMESLFGGGVQLDLEGFTPVGNGKGMMNPSAGMMTDPYIPMGMDIHGMGYGYSNLDLGMNYGSWGLLGDVF" } } ], "modelSeeds": [ 2077 ], "dialect": "alphafold3", "version": 1 }
2、将蛋白的fasta格式转换成为AlphaFold3使用的json格式
我自己编写了一个python脚本 “af3-pro_pro2json.py”(下载连接)可以将fasta格式整理成为AlphaFold3的格式,脚本中modelSeeds默认设置为2077,如果你需要修改的话可以把这个seeds改为任意的值。
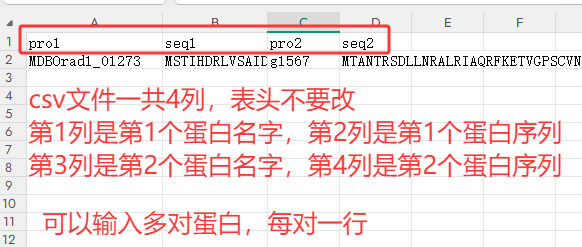
跟单个蛋白的结构预测不同,这里要预测的是两个蛋白的相互作用,因此我们的输入文件修改为一个csv文件,文件的格式如下:
整理好了csv文件,可以运行python脚本一次性生成json格式文件:
python3 af3-pro_pro2json.py proteins.csv
运行之后就会根据蛋白对的名称生成一系列的json格式文件,每对蛋白一个json格式文件。
3、进行蛋白结构预测
在当前目录下运行下面的代码,如果其他目录需要修改路径。
singularity exec \
--nv \
--bind ./af3-input:/root/af_input \
--bind ./af3-output:/root/af_output \
--bind ./af3-models:/root/models \
--bind ./af3-db:/root/public_databases \
alphafold3-new.sif \
python /app/alphafold/run_alphafold.py \
--input_dir=/root/af_input \
--model_dir=/root/models \
--db_dir=/root/public_databases \
--output_dir=/root/af_output
由于我们的服务器GPU只是4070 Ti Super,内存只有128 G,因此需要~50 min才能够预测完成,建议大家使用AI专用GPU或者至少5090以上的GPU。
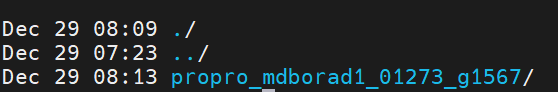
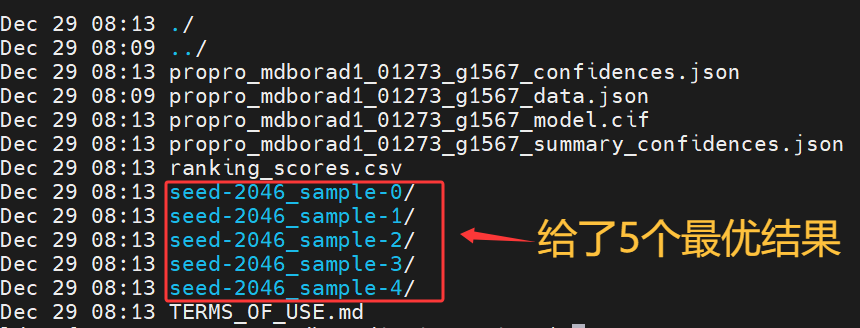
在af3-output目录下就会生成以json文件里面的name为名字的一个目录,里面放着结果文件。
进入目录之后可以看到结果,软件给出来了5个最优的结果,如果使用的序列和Seeds相同,那么生成的文件是完全一致的。
将这个文件下载到电脑上,用PyMOL等软件打开就可以直接看到结果。
二、利用AlphaFold3进行“蛋白-DNA/RNA”相互作用预测
(一)、AlphaFold3在线服务器进行 蛋白-核酸 互作分析
1、预测用的例子
这里预测用的例子直接从PDB数据库里面找,是一个经典的锌指蛋白Zif268的其中的最核心的功能片段。Zif268(又称 Egr1)由三个串联的 Cys_2His_2型锌指结构域(Finger 1, Finger 2, Finger 3)组成。每一个锌指结构域都通过 1 个锌离子 来稳定其核心结构(由两个半胱氨酸 Cys和两个组氨酸 His 协同配合)。核心片段就包含了这三个锌指结构域,因此有三个Zn原子。
蛋白和DNA复合物在PDB上面的编号为:1AAY 。通过下载的序列可以看到蛋白复合物包含3条链,前面2条分别是互补的DNA链,后面的1条是90 AA的蛋白核心结构域。
>1AAY_1|Chain A[auth B]|DNA (5’-D(APGPCPGPTPGPGPGPCPGPT)-3’)|
AGCGTGGGCGT
>1AAY_2|Chain B[auth C]|DNA (5’-D(TPAPCPGPCPCPCPAPCPGPC)-3’)|
TACGCCCACGC
>1AAY_3|Chain C[auth A]|PROTEIN (ZIF268 ZINC FINGER PEPTIDE)|Mus musculus (10090)
MERPYACPVESCDRRFSRSDELTRHIRIHTGQKPFQCRICMRNFSRSDHLTTHIRTHTGEKPFACDICGRKFARSDERKRHTKIHLRQKD
2、首先要打开AlphaFold3服务器,需要科学上网和Google账号。
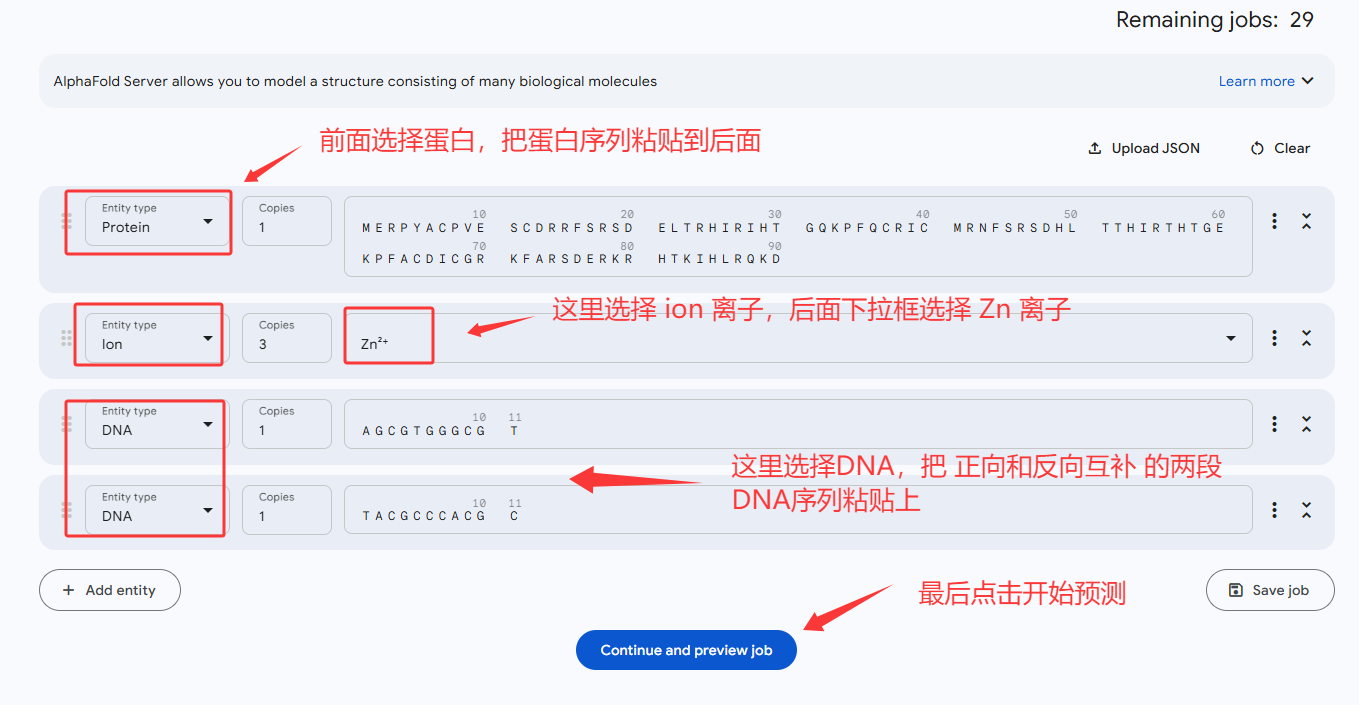
根据下图所示,以此把 蛋白序列、DNA序列、离子 给添加到要预测的结构复合物对话框中(顺序不重要)
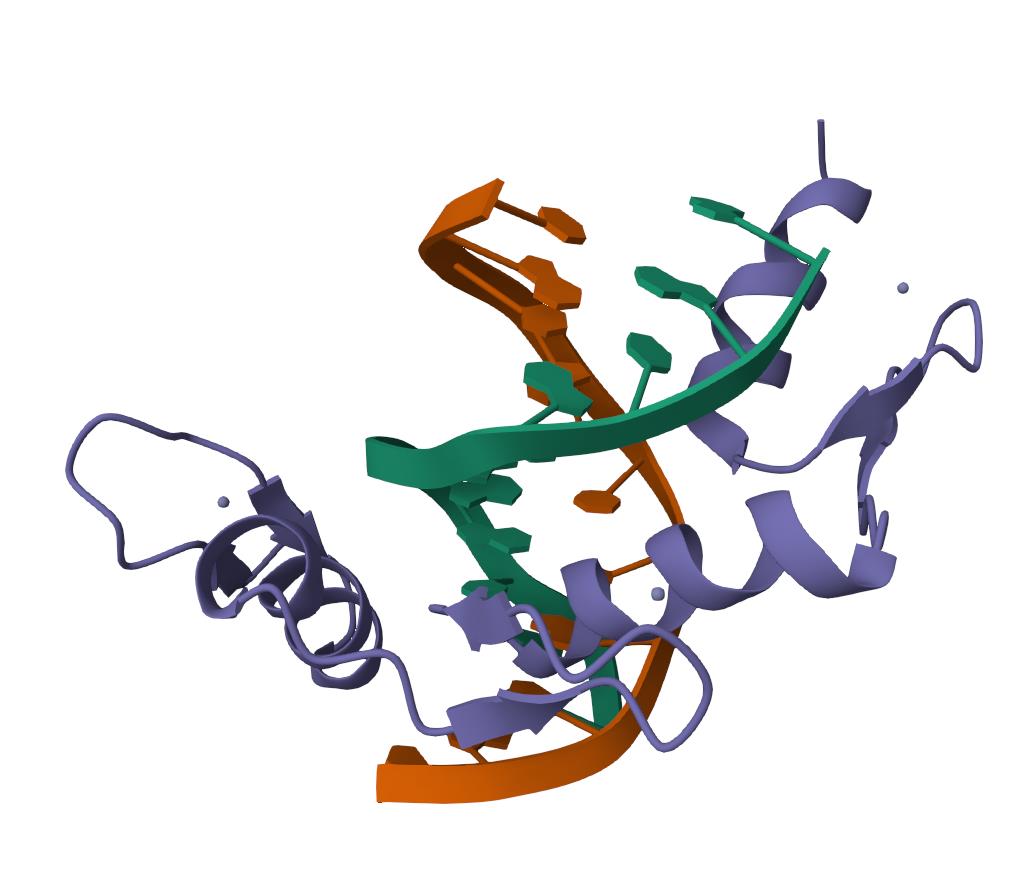
下面分别是AlphaFold3预测的结果和PDB数据库中给出来的晶体结构蛋白。
从结果来看,ipTM值为0.93,而且预测出来的蛋白复合物结构全部是深蓝色说明了预测准确性非常高。进一步跟PDB数据库中的晶体蛋白结构比较,虽然不能说两者如出一辙,但是也可以说是毫无二致了,AlphaFold3牛B。这个蛋白肯定是AlphaFold3模型训练的蛋白,所以说相似是必然的。
其他的蛋白和DNA的预测也可以参考上面的教程,应该记得把离子(ion)和配体(Ligond)都给加上。如果蛋白还有翻译后修饰(PTMs)也应该加上。
(二)、在本地Linux服务器进行“蛋白-核酸互作” 分析
运行脚本跟之前的完全一致,这里就把 JSON 格式给展示一下。
json格式的蛋白输入文件
{ "name": "prodna_zinc", "modelSeeds": [2077], "sequences": [ { "protein": { "id": "A", "sequence": "MERPYACPVESCDRRFSRSDELTRHIRIHTGQKPFQCRICMRNFSRSDHLTTHIRTHTGEKPFACDICGRKFARSDERKRHTKIHLRQKD" } }, { "ligand": { "id": ["Z", "Y", "X"], "ccdCodes": ["ZN"] } }, { "dna": { "id": ["E"], "sequence": "AGCGTGGGCGT" } }, { "dna": { "id": ["F"], "sequence": "TACGCCCACGC" } } ], "dialect": "alphafold3", "version": 1 }
利用ChimeraX、PyMOL等软件查看分析结果
在线服务器的结果可以下载到本地来查看,在线服务器会给出5个结果。本地运行的结果也是5个。
下载到本地的文件可以用PyMOL或者ChimeraX等软件进行查看并进行美化。
ChimeraX等软件的使用方法参考本网站其他教程



评论区